7. PCA#
7.1. Análisis#
Introducción
El Análisis de Componentes Principales (PCA) es un método para consolidar las variables mutuamente correlacionadas de datos observados multidimensionales en nuevas variables mediante combinaciones lineales de las variables originales con una pérdida mínima de información en los datos observados.
PCA permite extraer información relevante de los datos al fusionar múltiples variables que caracterizan a los individuos. También se puede utilizar como técnica para comprender visualmente las estructuras de datos al consolidar datos de alta dimensión en un número menor de variables, realizando una reducción de dimensión y proyectando los resultados en una línea unidimensional, plano bidimensional o espacio tridimensional.
En este capítulo se discuten los conceptos básicos y el propósito de PCA en el contexto de la linealidad, así como su aplicación en la descompresión de imágenes y la descomposición de valores singulares de matrices de datos. También se aborda el PCA no lineal utilizando el método del kernel para la extracción de información de datos multidimensionales con estructuras no lineales complejas.
7.2. Concepto básico#
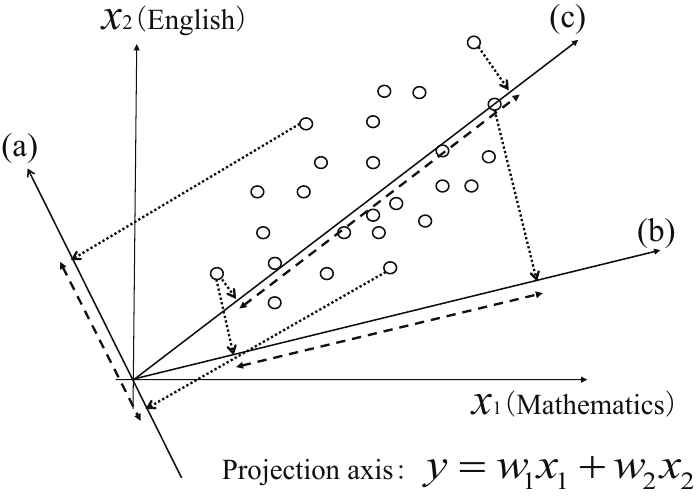
Fig. 7.1 Proyección en tres ejes diferentes, (a), (b) y (c), y la dispersión de los datos. Fuente [Konishi, 2014].#
La Fig. 7.1 muestra un gráfico de las puntuaciones de los exámenes de matemáticas (\(x_{1}\)) e inglés (\(x_{2}\)) de 25 estudiantes. Proyectando estos datos bidimensionales en un solo eje mediante la sustitución de las puntuaciones de ambas materias en la siguiente ecuación, se transforman en datos unidimensionales.
\[ y=w_{1}x_{1}+w_{2}x_{2}. \]
La Fig. 7.1 muestra la proyección en tres ejes diferentes, (a), (b) y (c). La dispersión de los datos (como medida de la varianza) para cada eje se indica mediante una flecha de doble cabeza (\(\boldsymbol{\leftarrow-\rightarrow}\)).
Observación
Esta figura muestra que la varianza es mayor cuando se proyecta en el eje (b) que en el eje (a), y mayor en el eje (c) que en el eje (b). El eje con mayor varianza muestra con mayor claridad la separación entre los datos. Surge la pregunta inmediata de cómo encontrar mejor el eje de proyección que produce la máxima varianza.
En PCA, utilizamos una secuencia de ejes de proyección con este propósito.
Primero encontramos el eje de proyección, conocido como el primer componente principal, que maximiza la varianza global.
Luego encontramos el eje de proyección que maximiza la varianza bajo la restricción de ortogonalidad al primer componente principal. Este eje se conoce como el segundo componente principal.
La ortogonalidad entre las componentes principales en PCA garantiza una representación óptima de la variabilidad de los datos, lo que facilita la interpretación y el análisis de los mismos.
Apliquemos lo mencionado en la anterior observación a datos bidimensionales. Denotamos los \(n\) datos observados bidimensionales \(\boldsymbol{x}=(x_{1}, x_{2})^{T}\) como
Estos \(n\) datos bidimensionales se proyectan sobre \(y=w_{1}x_{1}+w_{2}x_{2}\), y luego se expresan como
\[ y_{i}=w_{1}x_{i1}+w_{2}x_{i2}=\boldsymbol{w}^{T}\boldsymbol{x}_{i},~i=1,2,\dots,n, \]donde \(\boldsymbol{w}=(w_{1}, w_{2})^{T}\) representa un vector de coeficientes.
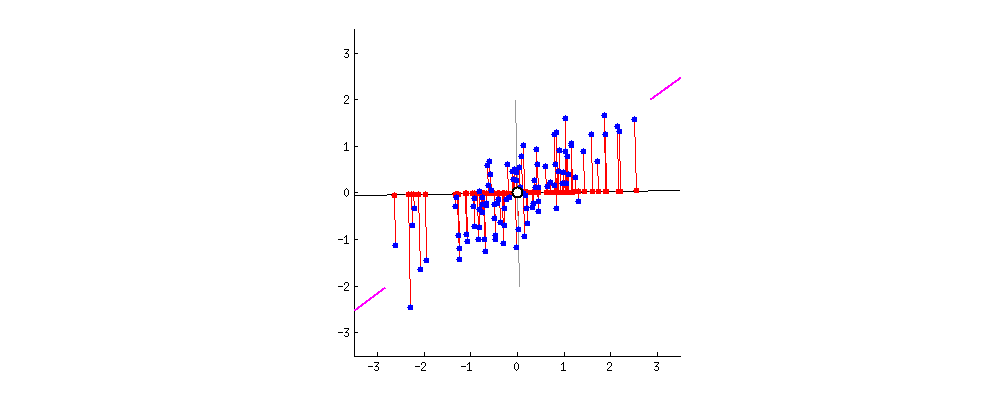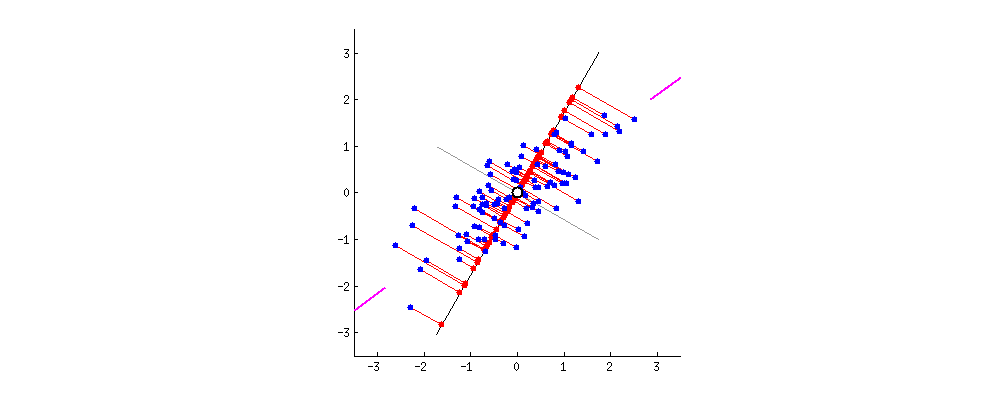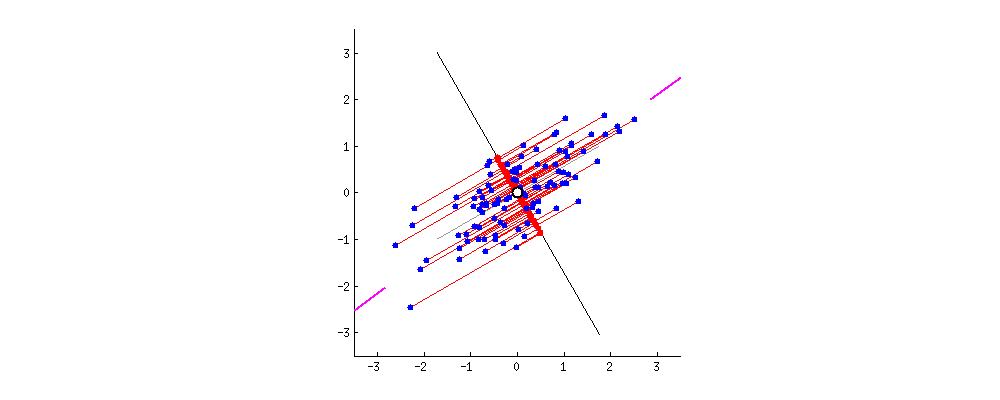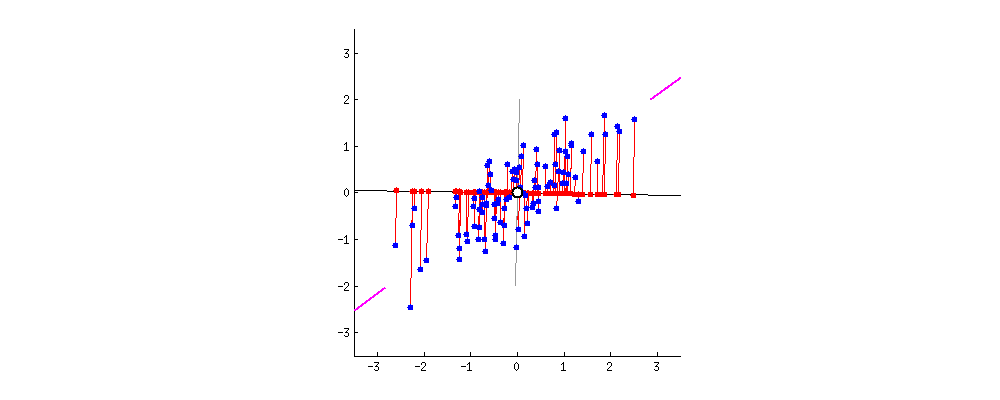
Fig. 7.2 Simulación de múltiples ejes de proyección \(y=w_{1}x_{1}+w_{2}x_{2}\) en el PCA.#
Datos proyectados repetidos?
Es posible que los datos proyectados en las componentes principales sean repetidos en ciertas circunstancias, especialmente cuando los datos originales tienen una alta dimensionalidad o cuando hay baja variabilidad en algunas dimensiones.
Esto es importante tener en cuenta al interpretar los resultados de PCA y al realizar análisis subsiguientes. Cuando esto ocurre, puedes considerar las siguientes estrategias:
Normalización de los datos
Reducción de multicolinealidad
Usar Análisis de Factores
La media de los datos \(y_{1}, y_{2}, \dots, y_{n}\) que se proyectan sobre el eje de proyección es
\[ \overline{y}=\frac{1}{n}\sum_{i=1}^{n}y_{i}=\frac{1}{n}\sum_{i=1}^{n}(w_{1}x_{i1}+w_{2}x_{i2})=w_{1}\overline{x}_{1}+w_{2}\overline{x}_{2}=\boldsymbol{w}\overline{\boldsymbol{x}}, \]donde \(\overline{\boldsymbol{x}}=(\overline{x}_{1}, \overline{x}_{2})^{T}\) es el vector de medias muestrales que contiene como sus componentes principales la media muestral \(\overline{x}_{j}=n^{-1}\sum_{i=1}^{n}x_{ij}~(j=1,2)\) de cada variable.
La varianza puede ser expresada como
(7.2)#\[\begin{split} \begin{align*} s_{y}^{2} &= \frac{1}{n}\sum_{i=1}^{n}(y_{i}-\overline{y})^{2}\\ &= \frac{1}{n}\sum_{i=1}^{n}\left\{w_{1}(x_{i1}-\overline{x}_{1})+w_{2}(x_{i2}-\overline{x}_{2})\right\}^{2}\\ &= w_{1}^{2}\frac{1}{n}\sum_{i=1}^{n}(x_{i1}-\overline{x}_{1})^{2}+2w_{1}w_{2}\frac{1}{n}\sum_{i=1}^{n}(x_{i1}-\overline{x}_{1})(x_{i2}-\overline{x}_{2})+w_{2}^{2}\frac{1}{n}\sum_{i=1}^{n}(x_{i2}-\overline{x}_{2})^{2}\\[1mm] &= w_{1}^{2}s_{11}+2w_{1}w_{2}s_{12}+w_{2}^{2}s_{22}\\[4mm] &= \boldsymbol{w}^{T}S\boldsymbol{w}, \end{align*} \end{split}\]donde \(S\) es la matriz de covarianza muestral, definida por (
verifíquelo)(7.3)#\[\begin{split} S=\begin{pmatrix}s_{11} & s_{12}\\s_{21} & s_{22}\end{pmatrix},~s_{jk}=\frac{1}{n}\sum_{i=1}^{n}(x_{ij}-\overline{x}_{j})(x_{ik}-\overline{x}_{k}),~j,k=1,2. \end{split}\]
Maximización de la varianza
El problema de encontrar el vector de coeficientes \(\boldsymbol{w}=(w_{1}, w_{2})^{T}\), que corresponde a la máxima varianza para los \(n\) datos bidimensionales proyectados sobre \(y=w_{1}x_{1}+w_{2}x_{2}\), se convierte en el problema de maximización de la varianza \(S_{y}=\boldsymbol{w}^{T}S\boldsymbol{w}\) en la Ecuación (7.2) bajo la restricción \(\boldsymbol{w}^{T}\boldsymbol{w}=1\). Esta restricción se aplica porque \(\|\boldsymbol{w}\|\) sería infinitamente grande sin ella y la varianza divergiría.
El problema de maximización de la varianza bajo esta restricción se puede resolver mediante el método de los multiplicadores de Lagrange, encontrando el punto estacionario (donde la derivada se hace 0) de la función Lagrangiana.
Dado que \(\partial(\boldsymbol{w}^{T}S\boldsymbol{w})/\partial\boldsymbol{w}=2S\boldsymbol{w}\) (ver Ecuación (3.9)) y \(\partial(\boldsymbol{w}^{T}\boldsymbol{w})/\partial\boldsymbol{w}=2\boldsymbol{w}\), entonces \(\partial L(\boldsymbol{w}, \lambda)/\partial\boldsymbol{w}=0\) sii
(7.4)#\[ S\boldsymbol{w}=\lambda\boldsymbol{w}. \]
Esta solución es el vector propio \(\boldsymbol{w}_{1}=(w_{11}, w_{12})^{T}\) correspondiente al valor propio máximo \(\lambda_{1}\) obtenido al resolver la ecuación característica para la matriz de varianza-covarianza de la muestra \(S\). Por consiguiente, el primer componente principal \(y_{1}\) está dado por
\[ y_{1}=w_{11}x_{1}+w_{12}x_{2}=\boldsymbol{w}_{1}^{T}\boldsymbol{x}. \]
La varianza del primer componente principal \(y_{1}\) se alcanza con \(\boldsymbol{w}=\boldsymbol{w}_{1}\), el cual maximiza la varianza de la Ecuación (7.2), y es, por lo tanto,
(7.5)#\[ s_{y_{1}}^{2}=\boldsymbol{w}_{1}^{T}S\boldsymbol{w}_{1}=\lambda_{1}. \]
Nótese que, la (7.5) es obtenida gracias a la relación entre autovalor-autovector \(S\boldsymbol{w}_{1}=\lambda_{1}\boldsymbol{w}_{1}\) de la matriz simétrica \(S\), y que el vector \(\boldsymbol{w}_{1}\) es normalizado a longitud 1 (
verifíquelo).
El segundo componente principal, al proyectar los datos bidimensionales en \(y=w_{1}x_{1}+w_{2}x_{2}\) bajo la restricción de normalización \(\boldsymbol{w}\boldsymbol{w}^{T}=1\) junto con la ortogonalidad al primer componente principal, se define como el vector de coeficientes que maximiza la varianza \(S_{y_{2}}^{2}=\boldsymbol{w}^{T}S\boldsymbol{w}\) con \(\boldsymbol{w}_{1}^{T}\boldsymbol{w}=0\).
La solución, de la misma manera que para el primer componente principal, se obtiene como el punto estacionario respecto a \(\boldsymbol{w}\) para la función Lagrangiana
\[ L(\boldsymbol{w}, \lambda, \gamma)=\boldsymbol{w}^{T}S\boldsymbol{w}+\lambda(1-\boldsymbol{w}^{T}\boldsymbol{w})+\gamma\boldsymbol{w}_{1}^{T}\boldsymbol{w}, \]donde \(\lambda, \gamma\) son multiplicadores de Lagrange.
Cuando diferenciamos parcialmente la ecuación anterior respecto a \(\boldsymbol{w}\) y establecemos el resultado igual a 0 (\(\partial_{\boldsymbol{w}}L=\boldsymbol{0}\)), obtenemos la ecuación
(7.6)#\[ \frac{\partial L(\boldsymbol{w},\lambda, \gamma)}{\partial\boldsymbol{w}}=0\Leftrightarrow 2S\boldsymbol{w}-2\lambda w+\gamma\boldsymbol{w}_{1}=\boldsymbol{0}. \]
Si luego multiplicamos a la izquierda por el vector propio \(\boldsymbol{w}_{1}^{T}\) correspondiente al valor propio máximo \(\lambda_{1}\), obtenemos
\[\begin{split} \begin{align*} 2\boldsymbol{w}_{1}^{T}S\boldsymbol{w}-2\lambda\textcolor{red}{\boldsymbol{w}_{1}^{T}\boldsymbol{w}}+\gamma\textcolor{red}{\boldsymbol{w}_{1}^{T}\boldsymbol{w}_{1}} &= 0\Leftrightarrow\begin{cases}\boldsymbol{w}_{1}^{T}\boldsymbol{w}&=0\\\boldsymbol{w}_{1}^{T}\boldsymbol{w}_{1}&=1\end{cases}\\ 2\textcolor{red}{\boldsymbol{w}_{1}^{T}S}\boldsymbol{w}+\gamma &= 0\Leftrightarrow (S\boldsymbol{w}_{1}=\lambda_{1}\boldsymbol{w}_{1}\Leftrightarrow\boldsymbol{w}_{1}^{T}S=\lambda_{1}\boldsymbol{w}_{1}^{T})\\[3mm] 2\textcolor{red}{\lambda\boldsymbol{w}_{1}^{T}}\boldsymbol{w}+\gamma &= 0\Leftrightarrow\\[3mm] \gamma &=0 \end{align*} \end{split}\]
Entonces la Ecuación (7.6) se convierte en
(7.7)#\[\begin{split} 2Sw-2\lambda\boldsymbol{w}+\gamma\boldsymbol{w}_{1}=2S\boldsymbol{w}-2\lambda\boldsymbol{2}=0\Leftrightarrow S\boldsymbol{w}=\lambda\boldsymbol{w},~\text{donde}~\begin{cases}\boldsymbol{w}_{1}^{T}\boldsymbol{w}&=0\\\boldsymbol{w}_{1}^{T}\boldsymbol{w}_{1}&=1\end{cases} \end{split}\]
La solución del problema de autovalores y autovectores asociado a la Ecuación (7.7) entrega el par autovector/autovalor \((\lambda_{2}, \boldsymbol{w}_{2})\), donde \(\boldsymbol{w}_{2}=(w_{21}, w_{22})^{T}\).
El segundo componente principal puede ser dado por la siguiente ecuación
\[ y_{2}=w_{21}x_{1}+w_{22}x_{2}=\boldsymbol{w}_{2}^{T}\boldsymbol{x} \]
De la relación entre el autovalor y el autovector de la matriz simétrica \(S\), la varianza del segundo componente principal \(y_{2}\) se convierte en \(s_{y_{2}}^{2}=\boldsymbol{w}_{2}S\boldsymbol{w}_{2}=\lambda_{2}\) de la misma manera que en la Ecuación (7.5).
El PCA basado en los datos bidimensionales en Ecuación (7.1) es esencialmente un problema de encontrar los autovalores y autovectores de la matriz de varianza-covarianza muestral \(S\).
Observación
Dado que cada componente principal
captura una parte única de la variabilidad en los datos, cada una contiene información diferente sobre la estructura de los datos originales. Esto significa que, mientras más componentes principales tengamos, más información detallada obtenemos sobre la distribución de los datos en el espacio de características.
7.3. Autovalores, Autovectores y Componentes Principales#
Denotamos los autovalores de la matriz de varianza-covarianza muestral \(S\) en orden descendente según su magnitud como \(\lambda_{1}\geq\lambda_{2}\geq0\), y los correspondientes autovectores mutuamente ortogonales normalizados a longitud 1 como \(\boldsymbol{w}_{1}=(w_{11}, w_{12})^{T}\) y \(\boldsymbol{w}_{2}=(w_{21}, w_{22})^{T}\).
Los primeros y segundos componentes principales y sus respectivas varianzas se dan entonces de la siguiente manera
\[\begin{split} \begin{align*} y_{1} &= w_{11}x_{1}+w_{12}x_{2},\quad\text{var}(y_{1})=\lambda_{1}\\ y_{2} &= w_{21}x_{1}+w_{22}x_{2},\quad\text{var}(y_{2})=\lambda_{2} \end{align*} \end{split}\]
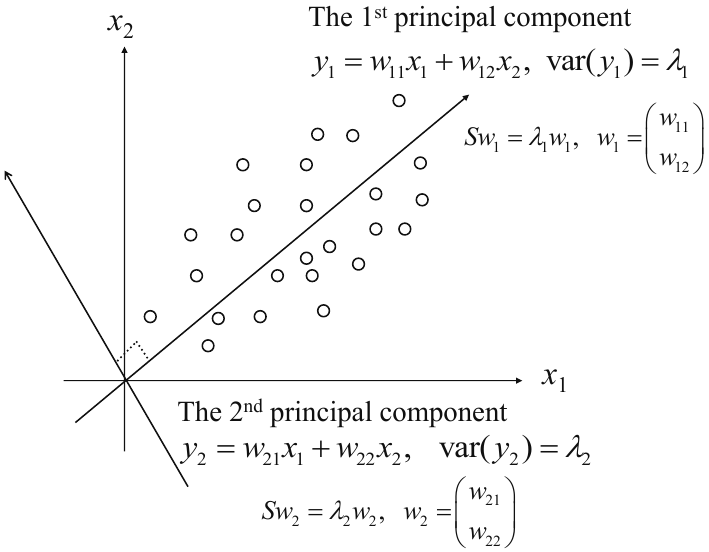
Fig. 7.3 Valores propios, primeros y segundos componentes principales. Fuente [Konishi, 2014].#
El método de los multiplicadores de Lagrange, como se utiliza en esta sección, proporciona una herramienta para encontrar los puntos estacionarios de funciones multivariables de valores reales bajo restricciones de igualdad y desigualdad en las variables.
7.4. Proceso de Derivación de Componentes Principales y Propiedades#
En general, denotamos como \(\boldsymbol{x}=(x_{1}, x_{2},\dots,x_{p})^{T}\) las \(p\) variables representando las características individuales. Basados en los datos observados \(\boldsymbol{x}_{1}, \boldsymbol{x}_{2},\dots, \boldsymbol{x}_{n}\); \(p\)-dimensionales, para las \(p\) variables, obtenemos la matriz de varianza-covarianza muestral
(7.8)#\[ S=(s_{jk})=\frac{1}{n}\sum_{i=1}^{n}(\boldsymbol{x}_{i}-\overline{\boldsymbol{x}})(\boldsymbol{x}_{i}-\boldsymbol{x})^{T} \]donde \(\overline{\boldsymbol{x}}\) es el vector de medias muestrales, \(p\)-dimensional, y \(s_{jk}=\sum_{i=1}^{n}(x_{ij}-\overline{x}_{j})(x_{ik}-\overline{x}_{k})/n\) (
escriba cada componente de\(S\)en forma matricial).
Siguiendo el concepto básico de la derivación de componentes principales, como se describe para datos bidimensionales en la sección anterior, primero proyectamos los \(n\) datos observados, \(p\)-dimensionales sobre el eje de proyección
(7.9)#\[ y=w_{1}x_{1}+w_{2}x_{2}+\cdots+w_{p}x_{p}=\boldsymbol{w}^{T}\boldsymbol{x}, \]y obtenemos los datos unidimensionales \(y_{i}=\boldsymbol{w}^{T}\boldsymbol{x}_{i}~(i=1,2,\dots,n;~\boldsymbol{x}_{i}~p\text{-dimensional})\).
La media de los datos proyectados, representada por \(\overline{y}=n^{-1}\sum_{i=1}^{n}\boldsymbol{w}^{T}\boldsymbol{x}_{i}=\boldsymbol{w}^{T}\overline{\boldsymbol{x}}\) (
escriba el vector de componentes de\(\overline{\boldsymbol{x}}\)) permite expresar la varianza como:\[\begin{split} \begin{align*} s_{y}^{2} &= \frac{1}{n}\sum_{i=1}^{n}(y_{i}-\overline{y})^{2}\\ &= \frac{1}{n}\sum_{i=1}^{n}(\boldsymbol{w}^{T}\boldsymbol{x}_{i}-\boldsymbol{w}^{T}\overline{\boldsymbol{x}})^{2}\\ &= \frac{1}{n}\sum_{i=1}^{n}(\boldsymbol{w}^{T}(\boldsymbol{x}_{i}-\overline{\boldsymbol{x}}))(\boldsymbol{w}^{T}(\boldsymbol{x}_{i}-\overline{\boldsymbol{x}}))^{T}\\ &= \boldsymbol{w}^{T}\frac{1}{n}\sum_{i=1}^{n}(\boldsymbol{x}_{i}-\overline{\boldsymbol{x}})(\boldsymbol{x}_{i}-\overline{\boldsymbol{x}})^{T}\boldsymbol{w}\\ &= \boldsymbol{w}^{T}S\boldsymbol{w}. \end{align*} \end{split}\]
De manera similar a lo aplicado en dos dimensiones, el vector de coeficientes que maximiza la varianza de los datos proyectados puede expresarse como el autovector \(\boldsymbol{w}_{1}\) correspondiente al mayor autovalor \(\lambda_{1}\) de la matriz de varianza-covarianza muestral \(S\).
El eje de proyección \(y_{1}=\boldsymbol{w}_{1}^{T}\boldsymbol{x}\) con \(\boldsymbol{w}_{1}\) como vector de coeficientes es entonces el primer componente principal. De la misma manera, nuevamente, la varianza en este eje de proyección es el mayor autovalor \(\lambda_{1}\)
El segundo componente principal es el eje que, cumpliendo con la ortogonalidad al primer componente principal, maximiza la varianza de los datos proyectados \(p\)-dimensionales, y, por lo tanto, es el eje de proyección generado por el autovector \(\boldsymbol{w}_{2}\), que corresponde al segundo mayor autovalor \(\lambda_{2}\) de la matriz \(S\)
Observación
Continuando de la misma manera, el tercer componente principal se define como el eje que, cumpliendo con la ortogonalidad al primer y segundo componente principal, maximiza la varianza de los datos proyectados \(p\)-dimensionales. Repitiendo sucesivamente este proceso, podemos en principio derivar \(p\) componentes principales para las combinaciones lineales de las variables originales.
La invertibilidad de la matriz de covarianza en PCA se garantiza debido a la necesidad de que las variables originales sean linealmente independientes, lo que conduce a una matriz de covarianza no singular.
Como resultado, PCA se convierte así en el problema de los autovalores de la matriz de varianza-covarianza muestral \(S\), como se describe a continuación.
7.4.1. El problema de los autovalores de la matriz de varianza-covarianza muestral y los componentes principales#
Denotemos \(S\) como una matriz de varianza-covarianza muestral basada en \(n\) datos observados \(p\)-dimensionales. Como se observa en la Ecuación (7.8), es una matriz simétrica de orden \(p\). Luego, denotamos los \(p\) autovalores
\[ \lambda_{1}\geq\lambda_{2}\geq\cdots\lambda_{i}\geq\cdots\geq\lambda_{p}\geq0, \]dados como la solución a la ecuación característica de \(S,~|S-\lambda I_{p}|=0\).
Además, denotamos los autovectores \(p\)-dimensionales normalizados a longitud 1 correspondientes a estos autovalores como
\[\begin{split} \boldsymbol{w}_{1}= \begin{pmatrix} w_{11}\\ w_{12}\\ \vdots\\ w_{1p} \end{pmatrix},~ \boldsymbol{w}_{2}= \begin{pmatrix} w_{21}\\ w_{22}\\ \vdots\\ w_{2p} \end{pmatrix},\dots, \boldsymbol{w}_{p}= \begin{pmatrix} w_{p1}\\ w_{p2}\\ \vdots\\ w_{pp} \end{pmatrix} \end{split}\]
Para estos autovectores, la normalización \(\boldsymbol{w}_{i}^{T}\boldsymbol{w}_{i}=1\) para longitud del vector 1 y la ortogonalidad \(\boldsymbol{w}_{i}^{T}\boldsymbol{w}_{j}=0\) (para \(i\neq j\)) quedan así establecidas.
Los \(p\) componentes principales y su varianza expresada en términos de la combinación lineal de las variables originales pueden ser proporcionados en orden de la siguiente manera
(7.10)#\[\begin{split} \begin{align*} y_{1} &= w_{11}x_{1}+w_{12}x_{2}+\cdots+w_{1p}x_{p}=\boldsymbol{w}_{1}^{T}\boldsymbol{x},\quad\text{var}(y_{1})=\lambda_{1},\\ y_{2} &= w_{21}x_{1}+w_{22}x_{2}+\cdots+w_{2p}x_{p}=\boldsymbol{w}_{2}^{T}\boldsymbol{x},\quad\text{var}(y_{2})=\lambda_{2},\\ &\vdots\\ y_{p} &= w_{p1}x_{1}+w_{p2}x_{2}+\cdots+w_{pp}x_{p}=\boldsymbol{w}_{p}^{T}\boldsymbol{x},\quad\text{var}(y_{p})=\lambda_{p}. \end{align*} \end{split}\]
Reducción de dimensión
Al aplicar \(PCA\), es posible reducir la dimensionalidad de los \(n\) datos observados \(p\)-dimensionales
\[ \{\boldsymbol{x}_{i}=(x_{i1}, x_{i2}, \dots, x_{ip})^{T}:~i=1,2,\dots, n\} \]para las \(p\) variables originales a un número menor, utilizando solo los primeros componentes principales; por ejemplo, a datos bidimensionales
\[ \{(y_{i1}, y_{i2}):~i=1,2,\dots,n\} \]utilizando solo el primero y el segundo componente principal, donde \(y_{i1}=\boldsymbol{w}_{1}^{T}\boldsymbol{x}_{i}\), \(y_{i2}=\boldsymbol{w}_{2}^{T}\boldsymbol{x}_{i}\)
Al proyectar así el conjunto de datos del espacio de dimensiones superior sobre un plano bidimensional, podemos aprender visualmente la estructura de los datos. Al encontrar el significado de las nuevas variables combinadas como combinaciones lineales de las variables originales, además, podemos extraer información útil.
El significado de los componentes principales puede entenderse en términos de la magnitud y el signo de los coeficientes \(w_{ij}\) de cada variable. Además, la correlación entre los componentes principales y las variables como indicador cuantitativo es muy útil para identificar las variables que influyen en los componentes principales. La correlación entre el \(i\)-ésimo componente principal \(y_{i}\) y la variable \(x_{j}\) está dada por
\[ r_{y_{i}, x_{j}}=\frac{\text{cov}(y_{i}, x_{j})}{\sqrt{\text{var}(y_{i})}\sqrt{\text{var}(x_{j})}}= \frac{\lambda_{i}w_{ij}}{\sqrt{\lambda_{i}}\sqrt{s_{jj}}}=\frac{\sqrt{\lambda_{i}}w_{ij}}{\sqrt{s_{jj}}} \]donde \(\lambda_{i}\) es la varianza del \(i\)-ésimo componente principal, \(w_{ij}\) es el coeficiente de la variable \(x_{j}\) para el \(i\)-ésimo componente principal, y \(s_{jj}\) es la varianza de la variable \(x_{j}\).
Observación (Reducción de dimensionalidad)
¿Cuántas componentes principales son necesarias?: Trazar un gráfico de la varianza explicada acumulativa en función del número de componentes y seleccionar el número de componentes que capturan una cantidad significativa de la varianza total (por ejemplo, el70% o el 80%).El 70% o 80% de varianza explicada es un umbral comúnmente utilizado en PCA para determinar el número de componentes principales que se deben retener en un modelo. Al retener el 70% o 80% de la varianza, se conserva la mayor parte de la información importante en los datos originales, y generalmente, conservar este porcentaje implica una reducción relevante en la dimensionalidad de los datos.
7.5. Autovalores y autovectores de una matriz simétrica#
La matriz de varianza-covarianza muestral \(S\) dada por la Ecuación (7.8) es una matriz simétrica de orden \(p\), con la siguiente relación entre sus autovalores y autovectores
(7.11)#\[ S\boldsymbol{w}_{i}=\lambda_{i}\boldsymbol{w}_{i},~\boldsymbol{w}_{i}^{T}\boldsymbol{w}_{i}=1,~\boldsymbol{w}_{i}^{T}\boldsymbol{w}_{j}=0~(i\neq j) \]para \(i,j=1,2,\dots,p\).
Denotamos como \(W\) la matriz de orden \(p\) que tiene como columnas los \(p\) autovectores, y como \(\Lambda\) la matriz de orden \(p\) que tiene los autovalores como sus elementos diagonales, es decir,
\[\begin{split} W=(\boldsymbol{w}_{1}, \boldsymbol{w}_{2}, \dots, \boldsymbol{w}_{p}),~ \Lambda = \begin{pmatrix} \lambda_{1} & 0 & \cdots & 0\\ 0 & \lambda_{2} & \cdots & 0\\ \vdots & \vdots & \ddots & \vdots\\ 0 & 0 & \vdots & \lambda_{p} \end{pmatrix} \end{split}\]
La relación entre los autovalores y autovectores de la matriz de varianza-covarianza muestral \(S\) dada por Ecuación (7.11) puede expresarse de la siguiente manera:
\(SW=W\Lambda,\quad W^{T}W=I_{p}\)
\(W^{T}SW=\Lambda\)
\(S=W\Lambda W^{T}=\lambda_{1}\boldsymbol{w}_{1}\boldsymbol{w}_{1}^{T}+\lambda_{2}\boldsymbol{w}_{2}\boldsymbol{w}_{2}^{T}+\cdots+\lambda_{p}\boldsymbol{w}_{p}\boldsymbol{w}_{p}^{T}\)
\(\text{tr}(S)=\text{tr}(W\Lambda W^{T})=\text{tr}(\Lambda)=\lambda_{1}+\lambda_{2}+\cdots+\lambda_{p}\)
Observación
La Ecuación (2) muestra que la matriz simétrica \(S\) puede ser diagonalizada por la matriz ortogonal \(W\), y la Ecuación (3) es conocida como la descomposición espectral de la matriz simétrica \(S\).
La Ecuación (4) muestra que la suma, \(\text{tr}(S)=s_{11}+s_{22}+\cdots+s_{pp}\), de las varianzas de las variables originales \(x_{1}, x_{2}, \dots, x_{p}\) es igual a la suma, \(\text{tr}(\Lambda)=\lambda_{1}+\lambda_{2}+\cdots+\lambda_{p}\), de las varianzas de los \(p\) componentes principales construidos.
7.6. Estandarización y matriz de correlación muestral#
Introducción
Supongamos que, además de las puntuaciones en matemáticas, inglés, ciencias y japonés, agregamos como quinta variable la proporción de respuestas correctas dadas para 100 problemas de cálculo que miden la habilidad para calcular y realizamos PCA basado en los datos resultantes de cinco dimensiones.
En este caso, las unidades de medida difieren sustancialmente entre la proporción de respuestas correctas y las puntuaciones de las pruebas de materias, por lo que es necesario estandarizar los datos observados.
Para los \(n\) datos observados \(p\) dimensionales
(7.12)#\[ \boldsymbol{x}_{i}=(x_{i1}, x_{i2}, \dots, x_{ip})^{T},\quad i=1, 2, \dots, n, \]Primero obtenemos el vector de media muestral \(\overline{\boldsymbol{x}}=(\overline{x}_{1}, \overline{x}_{2}, \dots, \overline{x}_{p})^{T}\) y la matriz de varianza-covarianza muestral \(S=(s_{jk})\). Estandarizamos los datos \(p\)-dimensionales en Ecuación (7.12) de manera que
\[ \boldsymbol{z}_{i}=(z_{i1}, z_{i2}, \dots, z_{ip})^{T},\quad z_{ij}=\frac{x_{ij}-\overline{x}_{j}}{\sqrt{s_{jj}}},\quad j=1, 2, \dots, p. \]
La varianza muestral (\(s_{jj}^{\star}\)) y la covarianza muestral (\(s_{jk}^{\star}\)) basadas en estos datos \(p\)-dimensionales estandarizados se dan entonces por
\[ s_{jk}^{\star}=\frac{1}{n}\sum_{i=1}^{n}z_{ij}z_{ik}=\frac{1}{n}\sum_{i=1}^{n}\frac{(x_{ij}-\overline{x}_{j})(x_{ik}-\overline{x}_{k})}{\sqrt{s_{jj}}\sqrt{s_{kk}}}=\frac{s_{jk}}{\sqrt{s_{jj}}\sqrt{s_{kk}}}\equiv r_{jk} \]para \(j, k=1,2,\dots,p\).
Observación
Nótese que en la matriz de varianza-covarianza muestral basada en los datos estandarizados, todos los elementos diagonales \(r_{jj}\) son 1, y obtenemos la matriz \(R\) con \(r_{jk}\) como los elementos no diagonales, los coeficientes de correlación muestral entre las variables \(j\)-ésima y \(k\)-ésima (matriz de correlación muestral).
PCA comenzando con datos multidimensionales estandarizados se convierte así en un problema de encontrar los autovalores y autovectores de la matriz de correlación muestral \(R\), lo cual se realiza mediante el mismo proceso de derivación de componentes principales que el de PCA comenzando con una matriz de varianza-covarianza muestral.
7.7. Reducción de dimensión y pérdida de información#
Introducción
En \(PCA\), la varianza proporciona una medida de información, y, por lo tanto, la pérdida de información puede ser estimada cuantitativamente a partir de los tamaños relativos de las varianzas de los componentes principales.
Como se muestra en Ecuación (7.10), la varianza del componente principal está dada por el autovalor de la matriz de varianza-covarianza muestral.
En resumen, podemos utilizar \(\lambda_{1}/(\lambda_{1}+\lambda_{2}+\cdots+\lambda_{p})\) para evaluar qué proporción de la información contenida en las \(p\) variables originales está presente en el primer componente principal \(y_{1}\).
En general, la siguiente ecuación se utiliza como medida de la información presente en el \(i\)-ésimo componente principal \(y_{i}\)
\[ \frac{\lambda_{i}}{\lambda_{1}+\lambda_{2}+\cdots+\lambda_{p}}. \]
De manera similar, el porcentaje de varianza explicada por los primeros \(k\) componentes principales se calcula mediante:
Observación
En conclusión, la medida cuantitativa de la información contenida en los componentes principales que realmente se utilizan se da por la relación de la suma de las varianzas de esos componentes principales a la suma de las varianzas de todos los componentes principales, lo que sirve como medida de la pérdida de información.
Example 7.1 (Relación entre estructura de correlación y pérdida de información)
Se analiza cómo la correlación entre variables afecta la varianza explicada por los componentes principales (PCA) en el caso bidimensional. Sean dos variables \(x_{1}\) y \(x_{2}\) con matriz de correlación muestral:
donde \(r (>0)\) es el coeficiente de correlación muestral entre \(x_{1}\) y \(x_{2}\). La diagonal contiene correlaciones iguales a uno porque cada variable está perfectamente correlacionada consigo misma, y \(R\) es simétrica y positiva semidefinida.
Ecuación característica. Para obtener los autovalores, se resuelve:
Las soluciones son
\(\lambda_1\) corresponde al primer componente principal (máxima varianza).
\(\lambda_2\) corresponde al segundo componente (varianza residual).
Cuando \(r \to 1\), se tiene \(\lambda_1 \to 2\) y \(\lambda_2 \to 0\), indicando que casi toda la varianza se concentra en un solo componente.
Autovectores. Para cada \(\lambda_i\), se resuelve \((R - \lambda_i I) \boldsymbol{w}_i = 0\). Las soluciones normalizadas son:
\(\boldsymbol{w}_1\) apunta en la dirección \(x_1 = x_2\) (ambas variables en conjunto).
\(\boldsymbol{w}_2\) es perpendicular, representando diferencias entre ellas.
Componentes principales. Proyectando los datos sobre los autovectores:
\(y_1\) captura la varianza compartida (información redundante).
\(y_2\) recoge la varianza no redundante.
Proporción de varianza explicada. Las fracciones de varianza explicada por cada componente son:
\(r\) |
Var(\(y_1\)) |
Var(\(y_2\)) |
|---|---|---|
0.0 |
0.50 |
0.50 |
0.5 |
0.75 |
0.25 |
0.9 |
0.95 |
0.05 |
A medida que \(r\) aumenta:
La varianza se concentra en el primer componente.
El segundo componente pierde relevancia informativa.
Por tanto, PCA puede reducir la dimensionalidad sin pérdida significativa cuando las variables están fuertemente correlacionadas.
Como se muestra en la Figura Fig. 7.4, la contribución del primer componente principal, y, por lo tanto, la proporción que puede ser explicada únicamente por el primer componente principal, aumenta con la correlación creciente entre las dos variables.
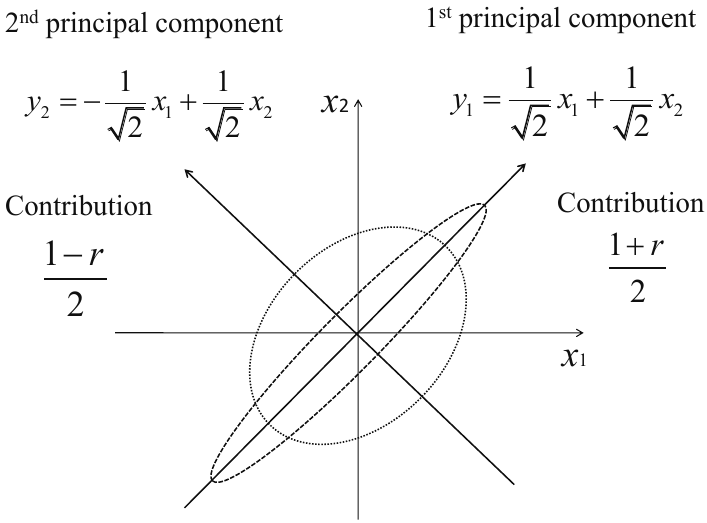
Fig. 7.4 Componentes principales basados en la matriz de correlación muestral y sus contribuciones. Fuente [Konishi, 2014].#
7.8. Implementación#
Hasta este momento, hemos analizado en detalle estimadores de aprendizaje supervisado: aquellos estimadores que predicen etiquetas basadas en datos de entrenamiento etiquetados. Ahora, empezamos a examinar varios estimadores no supervisados, los cuales pueden resaltar aspectos interesantes de los datos sin hacer referencia a etiquetas conocidas.
En esta sección, exploramos lo que posiblemente es uno de los algoritmos no supervisados más ampliamente utilizados: el análisis de componentes principales (PCA). PCA es fundamentalmente un algoritmo de reducción de dimensionalidad, pero también puede ser útil como herramienta de visualización, para filtrado de ruido, extracción y construcción de características, y mucho más.
import warnings
warnings.filterwarnings('ignore')
%matplotlib inline
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns; sns.set()
7.9. Introducción al análisis de componentes principales#
El análisis de componentes principales (PCA) es una técnica no supervisada, rápida y adaptable, diseñada para reducir la dimensionalidad de los datos. La forma más sencilla de comprender su funcionamiento es a través del examen de un conjunto de datos bidimensional. Tomemos en consideración la recopilación posterior de 200 puntos de datos
rng = np.random.RandomState(1)
X = np.dot(rng.rand(2, 2), rng.randn(2, 200)).T
plt.scatter(X[:, 0], X[:, 1])
plt.axis('equal');
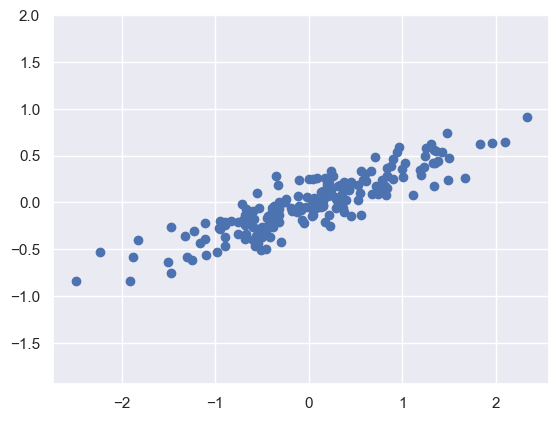
Visualmente, es evidente que
existe una conexión casi lineal entre las variables\(x\) e \(y\). En lugar de intentar predecir los valores de \(y\) basándose en los valores de \(x\), el objetivo delaprendizaje no supervisadoaquí escomprender la conexión entre los valoresde \(x\) e \(y\).El
análisis de componentes principales (PCA) cuantifica esta conexiónidentificando losejes principalesdentro de los datos yutilizándolos para representar el conjunto de datos. Para realizar esto utilizando el estimadorPCAdescikit-learn, siga estos pasos:
from sklearn.decomposition import PCA
pca = PCA(n_components=2)
pca.fit(X)
PCA(n_components=2)In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook.
On GitHub, the HTML representation is unable to render, please try loading this page with nbviewer.org.
PCA(n_components=2)
El proceso de ajuste
extrae información crucial de los datos, principalmente los"componentes"y la"varianza explicada".
print(pca.components_)
[[-0.94446029 -0.32862557]
[-0.32862557 0.94446029]]
print(pca.explained_variance_)
[0.7625315 0.0184779]
Para comprender el significado de estos valores, podemos
representarlos visualmente como vectores situados sobre los datos de entrada. En esta representación, los"componentes" determinan la dirección del vector, mientras que la"varianza explicada" determina el cuadrado de la longitud del vector.
def draw_vector(v0, v1, ax=None):
ax = ax or plt.gca()
arrowprops=dict(arrowstyle='->',
linewidth=2,
shrinkA=0, shrinkB=0, color='r')
ax.annotate('', v1, v0, arrowprops=arrowprops)
plt.scatter(X[:, 0], X[:, 1], alpha=0.5)
for length, vector in zip(pca.explained_variance_, pca.components_):
v = vector * 3 * np.sqrt(length)
draw_vector(pca.mean_, pca.mean_ + v)
plt.axis('equal');
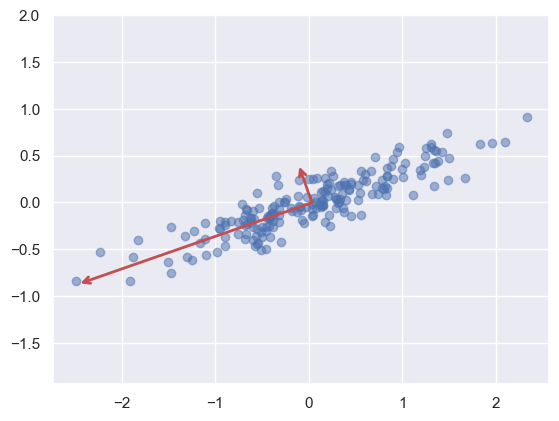
Observation 7.1
Estos vectores simbolizan las
orientaciones primarias de los datos, yla longitud del vectorindica laimportancia de esa orientación para explicar la distribución de los datos. Esta longitudcuantifica la varianza de los datos cuando se proyectan sobre esa orientación. La proyección de los puntos de datos sobre estas orientaciones principales se denomina"componentes principales"de los datos.
7.9.1. PCA para reducción de dimensión#
El
PCApuede emplearse parareducir la dimensionalidad anulando uno o varios de los componentes principales más pequeños. De este modo se obtiene unarepresentación de los datos de menor dimensión, conservando al mismo tiempo la mayor varianza posiblede los datos. A continuación se muestra un ejemplo de utilización dePCA comtransformación para la reducción de la dimensionalidad`
pca = PCA(n_components=1)
pca.fit(X)
X_pca = pca.transform(X)
print("original shape: ", X.shape)
print("transformed shape:", X_pca.shape)
original shape: (200, 2)
transformed shape: (200, 1)
Los
datos se han transformado y ahora existen en una sola dimensión. Paracomprender las implicaciones de esta reducciónde la dimensionalidad, podemos invertir el proceso de transformación en estos datos reducidos y luegocompararlos con los datos iniciales trazándolos juntos
X_new = pca.inverse_transform(X_pca)
plt.scatter(X[:, 0], X[:, 1], alpha=0.5)
plt.scatter(X_new[:, 0], X_new[:, 1], alpha=0.8)
plt.axis('equal');
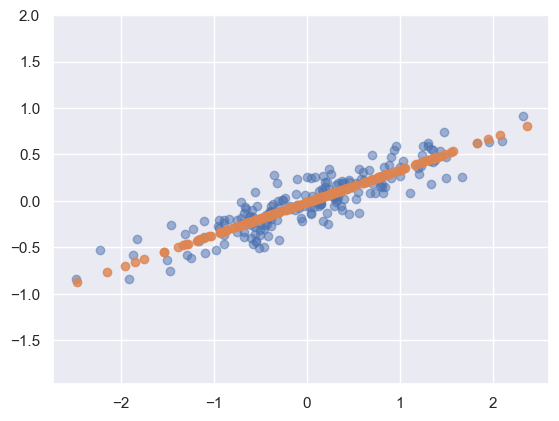
Los
puntos más claros en azulrepresentan los datos iniciales, mientras que losmás oscuros en salmonrepresentan laversión proyectada. Esta comparación visualaclara el concepto que subyace a la reducción de la dimensionalidad de PCA: consiste eneliminar los datos a lo largo del eje o ejes principales menos significativos, conservando únicamente el componente o componentes con mayor varianza.El
grado de reducción de la varianza(relacionado con la dispersión de los puntos alrededor de la línea en la figura) indica aproximadamente elgrado de "información" descartada en esta reducción de la dimensionalidad. A pesar de quela dimensionalidad se ha reducido a la mitad, este nuevo conjunto de datos es, en varios aspectos,suficientemente eficaz para captar las conexiones primarias entre los puntos de datos. Las relaciones fundamentales entre los puntos de datos se conservan en gran medida.
7.9.2. PCA para visualización: Dígitos escritos a mano#
El valor de
reducir la dimensionalidad puede no ser del todo evidente en sólo dos dimensiones, pero se hace mucho más evidente cuando se trata dedatos de alta dimensionalidad. Para ilustrarlo, examinaremos la aplicación dePCAal conjunto de datos dedígitos escritos a mano(imágenes \(8\times8\) (64-dimensional) de píxeles enteros en el rango \(0,\dots,16\).).
from sklearn.datasets import load_digits
from distinctipy import distinctipy
digits = load_digits(as_frame=True)
digits.data.shape
(1797, 64)
plt.matshow(digits.images[1]);
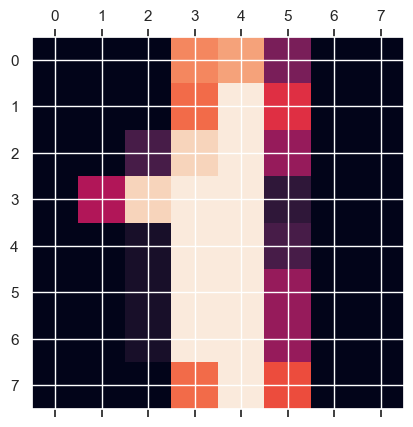
digits.images[1]
array([[ 0., 0., 0., 12., 13., 5., 0., 0.],
[ 0., 0., 0., 11., 16., 9., 0., 0.],
[ 0., 0., 3., 15., 16., 6., 0., 0.],
[ 0., 7., 15., 16., 16., 2., 0., 0.],
[ 0., 0., 1., 16., 16., 3., 0., 0.],
[ 0., 0., 1., 16., 16., 6., 0., 0.],
[ 0., 0., 1., 16., 16., 6., 0., 0.],
[ 0., 0., 0., 11., 16., 10., 0., 0.]])
digits.data.head()
| pixel_0_0 | pixel_0_1 | pixel_0_2 | pixel_0_3 | pixel_0_4 | pixel_0_5 | pixel_0_6 | pixel_0_7 | pixel_1_0 | pixel_1_1 | ... | pixel_6_6 | pixel_6_7 | pixel_7_0 | pixel_7_1 | pixel_7_2 | pixel_7_3 | pixel_7_4 | pixel_7_5 | pixel_7_6 | pixel_7_7 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 0.0 | 0.0 | 5.0 | 13.0 | 9.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | ... | 0.0 | 0.0 | 0.0 | 0.0 | 6.0 | 13.0 | 10.0 | 0.0 | 0.0 | 0.0 |
| 1 | 0.0 | 0.0 | 0.0 | 12.0 | 13.0 | 5.0 | 0.0 | 0.0 | 0.0 | 0.0 | ... | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 11.0 | 16.0 | 10.0 | 0.0 | 0.0 |
| 2 | 0.0 | 0.0 | 0.0 | 4.0 | 15.0 | 12.0 | 0.0 | 0.0 | 0.0 | 0.0 | ... | 5.0 | 0.0 | 0.0 | 0.0 | 0.0 | 3.0 | 11.0 | 16.0 | 9.0 | 0.0 |
| 3 | 0.0 | 0.0 | 7.0 | 15.0 | 13.0 | 1.0 | 0.0 | 0.0 | 0.0 | 8.0 | ... | 9.0 | 0.0 | 0.0 | 0.0 | 7.0 | 13.0 | 13.0 | 9.0 | 0.0 | 0.0 |
| 4 | 0.0 | 0.0 | 0.0 | 1.0 | 11.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | ... | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 2.0 | 16.0 | 4.0 | 0.0 | 0.0 |
5 rows × 64 columns
digits.data.describe()
| pixel_0_0 | pixel_0_1 | pixel_0_2 | pixel_0_3 | pixel_0_4 | pixel_0_5 | pixel_0_6 | pixel_0_7 | pixel_1_0 | pixel_1_1 | ... | pixel_6_6 | pixel_6_7 | pixel_7_0 | pixel_7_1 | pixel_7_2 | pixel_7_3 | pixel_7_4 | pixel_7_5 | pixel_7_6 | pixel_7_7 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| count | 1797.0 | 1797.000000 | 1797.000000 | 1797.000000 | 1797.000000 | 1797.000000 | 1797.000000 | 1797.000000 | 1797.000000 | 1797.000000 | ... | 1797.000000 | 1797.000000 | 1797.000000 | 1797.000000 | 1797.000000 | 1797.000000 | 1797.000000 | 1797.000000 | 1797.000000 | 1797.000000 |
| mean | 0.0 | 0.303840 | 5.204786 | 11.835838 | 11.848080 | 5.781859 | 1.362270 | 0.129661 | 0.005565 | 1.993879 | ... | 3.725097 | 0.206455 | 0.000556 | 0.279354 | 5.557596 | 12.089037 | 11.809126 | 6.764051 | 2.067891 | 0.364496 |
| std | 0.0 | 0.907192 | 4.754826 | 4.248842 | 4.287388 | 5.666418 | 3.325775 | 1.037383 | 0.094222 | 3.196160 | ... | 4.919406 | 0.984401 | 0.023590 | 0.934302 | 5.103019 | 4.374694 | 4.933947 | 5.900623 | 4.090548 | 1.860122 |
| min | 0.0 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | ... | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 |
| 25% | 0.0 | 0.000000 | 1.000000 | 10.000000 | 10.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | ... | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 1.000000 | 11.000000 | 10.000000 | 0.000000 | 0.000000 | 0.000000 |
| 50% | 0.0 | 0.000000 | 4.000000 | 13.000000 | 13.000000 | 4.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | ... | 1.000000 | 0.000000 | 0.000000 | 0.000000 | 4.000000 | 13.000000 | 14.000000 | 6.000000 | 0.000000 | 0.000000 |
| 75% | 0.0 | 0.000000 | 9.000000 | 15.000000 | 15.000000 | 11.000000 | 0.000000 | 0.000000 | 0.000000 | 3.000000 | ... | 7.000000 | 0.000000 | 0.000000 | 0.000000 | 10.000000 | 16.000000 | 16.000000 | 12.000000 | 2.000000 | 0.000000 |
| max | 0.0 | 8.000000 | 16.000000 | 16.000000 | 16.000000 | 16.000000 | 16.000000 | 15.000000 | 2.000000 | 16.000000 | ... | 16.000000 | 13.000000 | 1.000000 | 9.000000 | 16.000000 | 16.000000 | 16.000000 | 16.000000 | 16.000000 | 16.000000 |
8 rows × 64 columns
digits.data.isna().any().any()
False
color_bars = ['black', 'red', 'green', 'blue', 'cyan', 'purple', 'orange', 'pink', 'brown', 'yellow']
digits.target.value_counts().plot(kind='bar', color=distinctipy.get_colors(10));
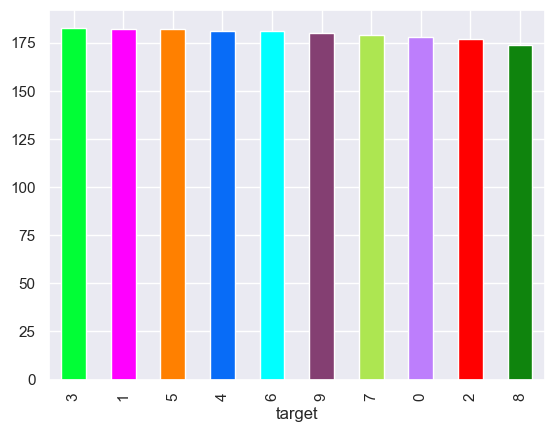
Para
comprender mejor las conexiones entre estos puntos, podemos utilizar elPCA para condensarlos en un número de dimensiones más manejable, por ejemplo,dos dimensiones (proyectar de 64 a 2 dimensiones)
import pandas as pd
pca = PCA(2)
projected = pca.fit_transform(digits.data)
print(digits.data.shape)
print(projected.shape)
(1797, 64)
(1797, 2)
El
trazado de los dos componentes principales inicialesde cada punto nos permiteextraer información de los datos.
plt.scatter(projected[:, 0], projected[:, 1],
c=digits.target, edgecolor='none', alpha=0.7,
cmap=plt.cm.get_cmap('viridis', 10));
plt.xlabel('PC 1');
plt.ylabel('PC 2');
plt.colorbar();
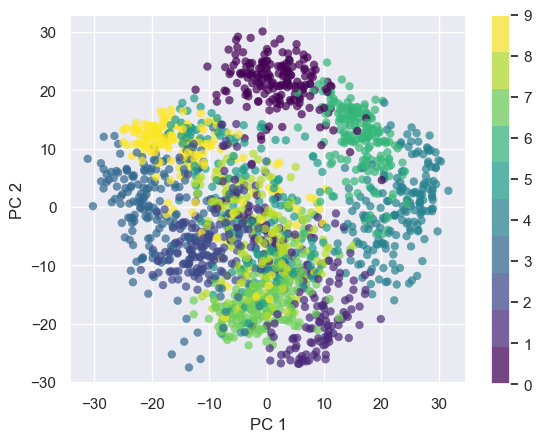
Observation 7.2
Consideremos la
importancia de estos componentes: el conjunto completo de datos forma unanube de puntos de 64 dimensiones, y estos puntos concretos representan laproyección de cada punto de datos a lo largo de las direcciones con mayor varianza.Fundamentalmente, hemos
identificado el estiramiento y la rotación más eficaces en el espacio de 64 dimensiones, lo que nos permiteobservar la disposición de los dígitos en dos dimensiones. Y lo que es más importante, esto se ha logradode forma no supervisada, sin utilizar ninguna etiqueta.
El PCA puede conceptualizarse como la selección de
funciones base óptimas. Lacombinación de sólo un pequeño subconjunto de estas funciones basta para reconstruir eficazmente la mayoría de los elementos del conjunto de datos. Los componentes principales, que sirven de representación condensada de los datos en dimensiones inferiores, son esencialmente loscoeficientes que escalan cada elemento de esta serie.PCAnos permiterecuperar las características más destacadas de la imagen de entrada¡con sólo una media más ocho componentes! La cantidad de cada píxel en cada componente es el corolario de laorientación del vector en nuestro ejemplo bidimensional.
7.9.3. Selección del número de componentes#
Un aspecto esencial de la aplicación práctica del
PCAconsiste endeterminar el número óptimo de componentes necesarios para caracterizar los datos. Esto puede lograrse analizando laproporción de varianza explicada acumulativa en relación con el número de componentes
pca = PCA().fit(digits.data)
plt.plot(np.cumsum(pca.explained_variance_ratio_))
plt.xlabel('number of components')
plt.ylabel('cumulative explained variance');
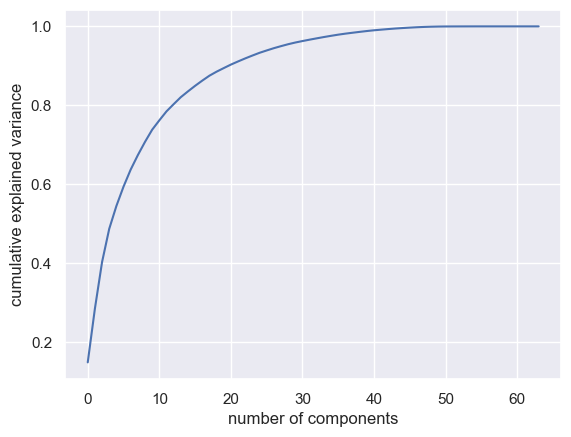
La curva presentada proporciona una
medida de qué parte de la varianza total, que abarca64 dimensiones, queda englobada por los \(N\)componentes iniciales. Por ejemplo, en elcaso de los dígitos, los 10 componentes iniciales representan aproximadamente el 75\% de la varianza, mientras que se necesitan unos50 componentes para describir casi el 100\% de la varianza.De ello se desprende que nuestra
proyección bidimensional conlleva una pérdida significativa de información, como demuestra la varianza explicada. Paraconservar aproximadamente el 90\% de la varianza, serían necesarios unos 20 componentes. El análisis de este gráfico para un conjunto de datos con dimensiones elevadas ayuda acomprender el grado de redundancia presente en numerosas observaciones.
7.9.4. PCA como filtro de ruido#
El PCA puede servir como técnica para
filtrar el ruido de los datos. El concepto fundamental es que loscomponentes con una varianza significativamente mayor que el impacto del ruido, están menos influidas por éste. Por consiguiente, si se reconstruyen los datos utilizando elsubconjunto primario de componentes principales con la mayor varianza, se conserva efectivamente la señal al tiempo que se elimina el ruido.Para observar esto en acción con el conjunto de datos de
dígitos, empezaremos generandográficos para varias instancias de los datos originales libres de ruido
digits = load_digits()
def plot_digits(data):
fig, axes = plt.subplots(4, 10, figsize=(10, 4),
subplot_kw={'xticks':[], 'yticks':[]},
gridspec_kw=dict(hspace=0.1, wspace=0.1))
for i, ax in enumerate(axes.flat):
ax.imshow(data[i].reshape(8, 8),
cmap='binary', interpolation='nearest', clim=(0, 16))
plot_digits(digits.data)
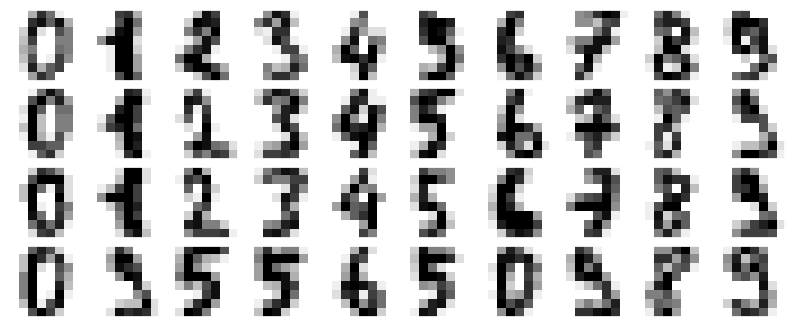
Ahora,
introduciremos ruido aleatorio para generar un conjunto de datos con ruidoy, a continuación,volveremos a crear los gráficos
np.random.seed(42)
noisy = np.random.normal(loc=digits.data, scale=4)
plot_digits(noisy)
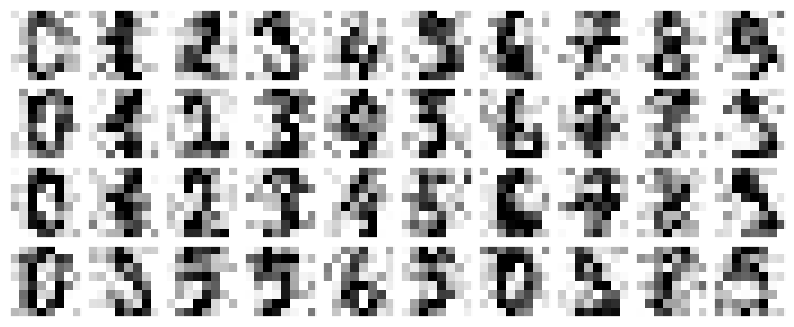
Las imágenes muestran un
ruido evidente e incluyen píxeles erróneos. Procederemos entrenando unPCA sobre los datos ruidosos, especificando quela proyección debe mantener el 50% de la varianza
pca = PCA(0.50).fit(noisy)
pca.n_components_
12
En este caso, el
50% de la varianza corresponde a 12 componentes principales. Para proceder,calculamos estos componentesy posteriormente utilizamos lainversa del proceso de transformación para reconstruir los dígitos filtrados
components = pca.transform(noisy)
filtered = pca.inverse_transform(components)
plot_digits(filtered)
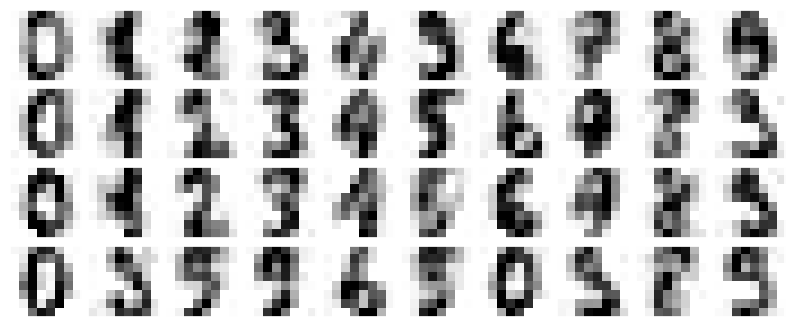
La
capacidad de PCA para retener señales y filtrar el ruido la convierte en una técnica muy valiosa para la selección de características. Por ejemplo,en lugar de entrenar un clasificador en datos con numerosas dimensiones, se puede optar porentrenar el clasificador en la representación de dimensiones reducidas, que elimina intrínsecamente el ruido aleatoriode las entradas.
7.9.5. Ejemplo Eigenfaces#
Examinamos la utilización de una
proyección PCAcomoselector de característicasen el contexto delreconocimiento facial. Profundicemos en los detalles, trabajado con el conjunto de datosLabeled Faces in the Wildproporcionado porscikit-learn
from sklearn.datasets import fetch_lfw_people
faces = fetch_lfw_people(min_faces_per_person=60)
print(faces.target_names)
print(faces.images.shape)
['Ariel Sharon' 'Colin Powell' 'Donald Rumsfeld' 'George W Bush'
'Gerhard Schroeder' 'Hugo Chavez' 'Junichiro Koizumi' 'Tony Blair']
(1348, 62, 47)
min_faces_per_person=60asegura que solo se incluyan personas con al menos 60 imágenes disponibles en el conjunto de datos.(1348, 62, 47)está asociado con el shape de las imágenes de los rostros en el conjunto de datos. Indica que hay 1348 imágenes en total. Cada imagen tiene dimensiones de 62 píxeles de alto por 47 píxeles de ancho. Esto significa que cada imagen tiene una resolución de 62x47 píxeles y que hay 1348 imágenes en total en el conjunto de datos.
A continuación
examinaremos los ejes principales que engloban este conjunto de datos. Debido a suconsiderable tamaño, emplearemosrandomized PCA. Este enfoque emplea unatécnica aleatoria para aproximar rápidamente los\(N\)componentes principales iniciales, lo que resulta especialmente ventajoso para datos de gran dimensión, como éste, con casi3.000 dimensiones. Nos centraremos en los primeros 150 componentes
pca = PCA(150, svd_solver='randomized')
pca.fit(faces.data)
PCA(n_components=150, svd_solver='randomized')In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook.
On GitHub, the HTML representation is unable to render, please try loading this page with nbviewer.org.
PCA(n_components=150, svd_solver='randomized')
En este caso, resulta interesante
representar visualmente las imágenes correspondientes a los componentes principales iniciales. Formalmente denominados"vectores propios", estos componentes suelen denominarse"caras propias" cuando se trata de imágenes. Como se muestra en la figura, estas caras propias tienen un aspecto bastante inquietante
fig, axes = plt.subplots(3, 8, figsize=(9, 4),
subplot_kw={'xticks':[], 'yticks':[]},
gridspec_kw=dict(hspace=0.1, wspace=0.1))
for i, ax in enumerate(axes.flat):
ax.imshow(pca.components_[i].reshape(62, 47), cmap='bone')

Nótese que las
caras propias iniciales(empezando por la parte superior izquierda) parecen estarrelacionadas con el ángulo de iluminación de la cara, mientras que losvectores principales posteriores empiezan a aislar rasgos específicos como los ojos, la nariz y los labios. Examinemos ahora lavarianza acumulada asociada a estos componentespara calibrar hasta qué punto la proyección conserva la información de los datos
plt.plot(np.cumsum(pca.explained_variance_ratio_))
plt.xlabel('number of components')
plt.ylabel('cumulative explained variance');
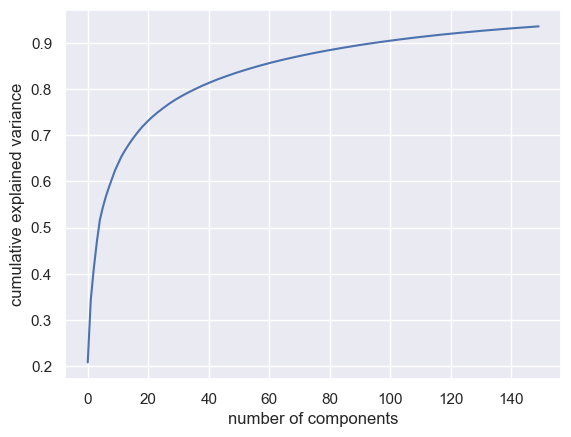
Se observa que estos
150 componentes abarcan algo más del 90% de la varianza. Esto sugiere que, utilizando estos 150 componentes,probablemente recuperaríamos la mayoría de los atributos críticos de los datos. Para consolidar esta idea, podemoscomparar las imágenes de entrada originales con las imágenes reconstruidas utilizando estos 150 componentes
pca = PCA(150, svd_solver='randomized').fit(faces.data)
components = pca.transform(faces.data)
projected = pca.inverse_transform(components)
fig, ax = plt.subplots(2, 10, figsize=(10, 2.5),
subplot_kw={'xticks':[], 'yticks':[]},
gridspec_kw=dict(hspace=0.1, wspace=0.1))
for i in range(10):
ax[0, i].imshow(faces.data[i].reshape(62, 47), cmap='binary_r')
ax[1, i].imshow(projected[i].reshape(62, 47), cmap='binary_r')
ax[0, 0].set_ylabel('full-dim\ninput')
ax[1, 0].set_ylabel('150-dim\nreconstruction');

Las imágenes de la
fila superior representan la entrada original, mientras que lafila inferior muestra imágenes reconstruidas utilizando sólo 150 de las aproximadamente 3.000 características iniciales. Esta visualización ilustra eficazmente por qué la selección de características PCA, logra un éxito significativo.A pesar de
reducir significativamente la dimensionalidadde los datos en aproximadamente un factor de 20,las imágenes proyectadas conservan suficiente información para que podamos reconocer a los individuosen las imágenes mediante inspección visual.Esto implica que nuestro
algoritmo de clasificación puede entrenarse con datos de 150 dimensiones en lugar de con datos de 3.000 dimensiones. Dependiendo del algoritmo específico elegido, esto puede conducir a unaclasificación mucho más eficiente.
