10. Pipelines#
El rendimiento de los algoritmos de aprendizaje automático depende de la representación de los datos, desde el escalado hasta el aprendizaje de características no supervisadas. La mayoría de las aplicaciones requieren encadenar múltiples transformaciones y modelos.
La clase
Pipelinesimplifica este proceso, permitiendo combinar pasos de preprocesamiento y modelos en un solo flujo. Además,Pipelinepuede integrarse conGridSearchCVpara optimizar todos los parámetros a la vez. Un ejemplo muestra que el uso deMinMaxScalermejora significativamente el desempeño de unSVMen el conjunto de datoscancer.
import numpy as np
from sklearn.linear_model import LogisticRegression
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
from sklearn.metrics import roc_auc_score
np.random.seed(0)
n = 1000
X = np.random.rand(n, 5)
y = np.random.randint(0, 2, size=n)
leaky_feature = y + np.random.normal(0, 0.001, size=n)
X_with_leak = np.hstack([X, leaky_feature.reshape(-1, 1)])
Primer intento usando el modelo de clasificación
LogisticRegression()
scaler = StandardScaler().fit(X_with_leak)
X_scaled = scaler.transform(X_with_leak)
X_train, X_test, y_train, y_test = train_test_split(X_scaled, y, test_size=0.3, random_state=42)
model = LogisticRegression()
model.fit(X_train, y_train)
y_scores = model.predict_proba(X_test)[:, 1]
print("AUC con data leakage: {:.3f}".format(roc_auc_score(y_test, y_scores)))
AUC con data leakage: 1.000
Segundo intento usando el modelo de clasificación
LogisticRegression()
X_clean = X
X_train, X_test, y_train, y_test = train_test_split(X_clean, y, test_size=0.3, random_state=42)
scaler = StandardScaler().fit(X_train)
X_train_scaled = scaler.transform(X_train)
X_test_scaled = scaler.transform(X_test)
model = LogisticRegression()
model.fit(X_train_scaled, y_train)
y_scores = model.predict_proba(X_test_scaled)[:, 1]
print("AUC sin data leakage: {:.3f}".format(roc_auc_score(y_test, y_scores)))
AUC sin data leakage: 0.499
10.1. Selección de parámetros con preprocesamiento#
Ahora digamos que queremos encontrar mejores parámetros para el
SVCusandoGridSearchCV, como se ha discutido en secciones anteriores. ¿Cómo podemos hacerlo? Un enfoque ingenuo podría ser el siguiente
import numpy as np
from sklearn.svm import SVC
from sklearn.pipeline import Pipeline
from sklearn.model_selection import train_test_split, GridSearchCV
from sklearn.preprocessing import MinMaxScaler
from sklearn.metrics import roc_auc_score
np.random.seed(0)
n = 1000
X = np.random.rand(n, 5)
y = np.random.randint(0, 2, size=n)
leaky_feature = y + np.random.normal(0, 0.001, size=n)
X_with_leak = np.hstack([X, leaky_feature.reshape(-1, 1)])
X_train, X_test, y_train, y_test = train_test_split(
X_with_leak, y, test_size=0.3, random_state=42
)
scaler = MinMaxScaler().fit(X_train)
X_train_scaled = scaler.transform(X_train)
X_test_scaled = scaler.transform(X_test)
param_grid = {
'C': [0.01, 0.1, 1, 10, 100],
'gamma': [0.01, 0.1, 1, 10, 100]
}
grid_leak = GridSearchCV(SVC(probability=True), param_grid=param_grid, cv=5, scoring="roc_auc")
grid_leak.fit(X_train_scaled, y_train)
y_scores_leak = grid_leak.predict_proba(X_test_scaled)[:, 1]
auc_leak = roc_auc_score(y_test, y_scores_leak)
print("AUC con data leakage (sin pipeline): {:.3f}".format(auc_leak))
print("Mejores parámetros (con leakage):", grid_leak.best_params_)
AUC con data leakage (sin pipeline): 1.000
Mejores parámetros (con leakage): {'C': 0.01, 'gamma': 0.01}
Se realiza una búsqueda en red (
grid-search) sobre los parámetros deSVCutilizando los datos escalados. Al escalar, se usa todo el conjunto de entrenamiento, y los datos escalados se emplean en la búsqueda en red con validación cruzada (cv=5). En cada división, una parte del conjunto de entrenamiento se usa para entrenar y otra para evaluar, simulando cómo el modelo clasificaría nuevos datos.Es clave notar que, en la validación cruzada, la parte de prueba sigue perteneciendo al conjunto de entrenamiento y el escalado se calcula con todos los datos de entrenamiento. Sin embargo, en la evaluación final con datos nuevos, estos pueden tener un mínimo y un máximo diferentes, afectando el escalado. El siguiente ejemplo ilustra este proceso en la validación cruzada y la evaluación final.
import mglearn
mglearn.plots.plot_improper_processing()
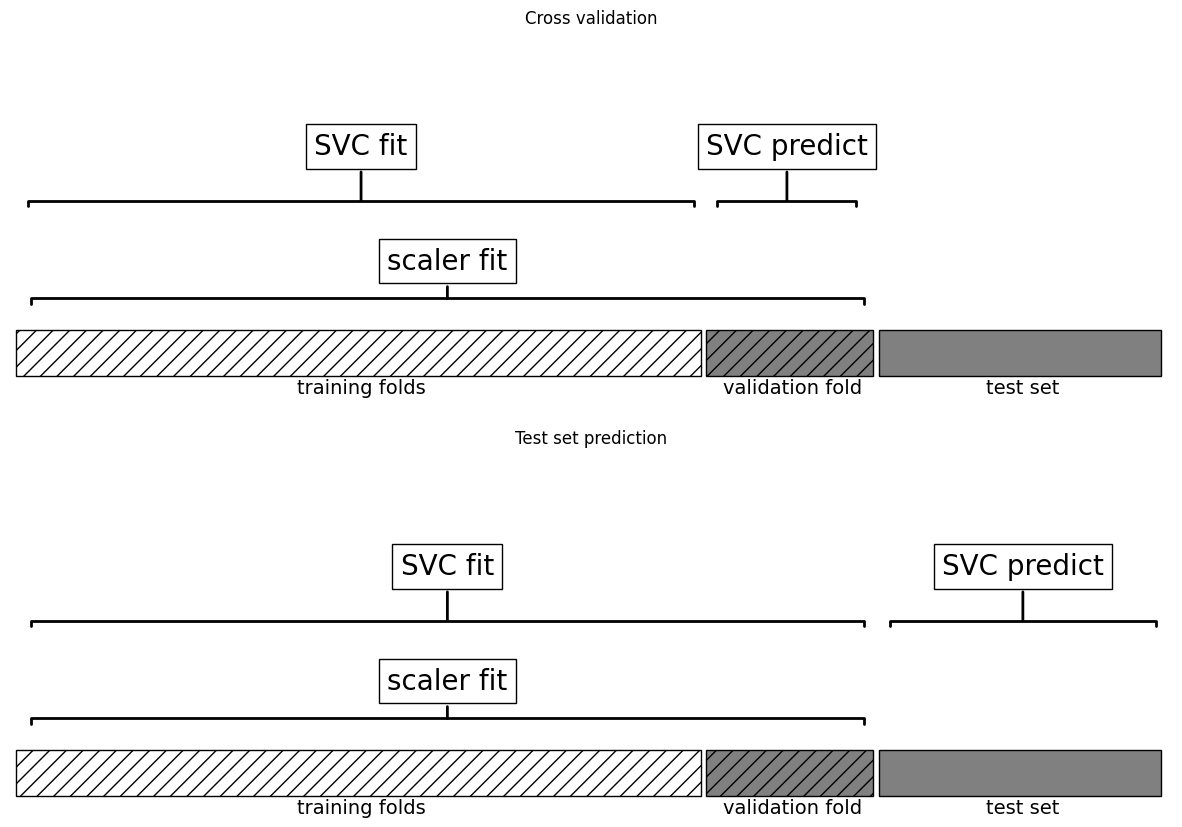
La validación cruzada puede dar resultados demasiado optimistas si se filtra información antes de la división de los datos, lo que puede llevar a la selección de parámetros subóptimos. Para evitarlo, la división debe hacerse antes del preprocesamiento, asegurando que cualquier conocimiento extraído solo afecte el conjunto de entrenamiento. La validación cruzada debe ser el “bucle más externo” del proceso.
En
scikit-learn, esto se maneja conPipeline, que permite combinar varios pasos de preprocesamiento y modelado en un solo estimador.Pipelinefacilita el ajuste, la predicción y la evaluación, integrando transformaciones como el escalado junto con modelos supervisados.
Observacion
La fuga de datos ocurre cuando el modelo usa información ajena al conjunto de entrenamiento, afectando su evaluación. Si hay datos compartidos entre entrenamiento y prueba, los resultados pueden ser artificialmente altos, ya que el modelo ha sido expuesto previamente a los datos de prueba.
10.2. Construyendo Pipelines with GridSearch#
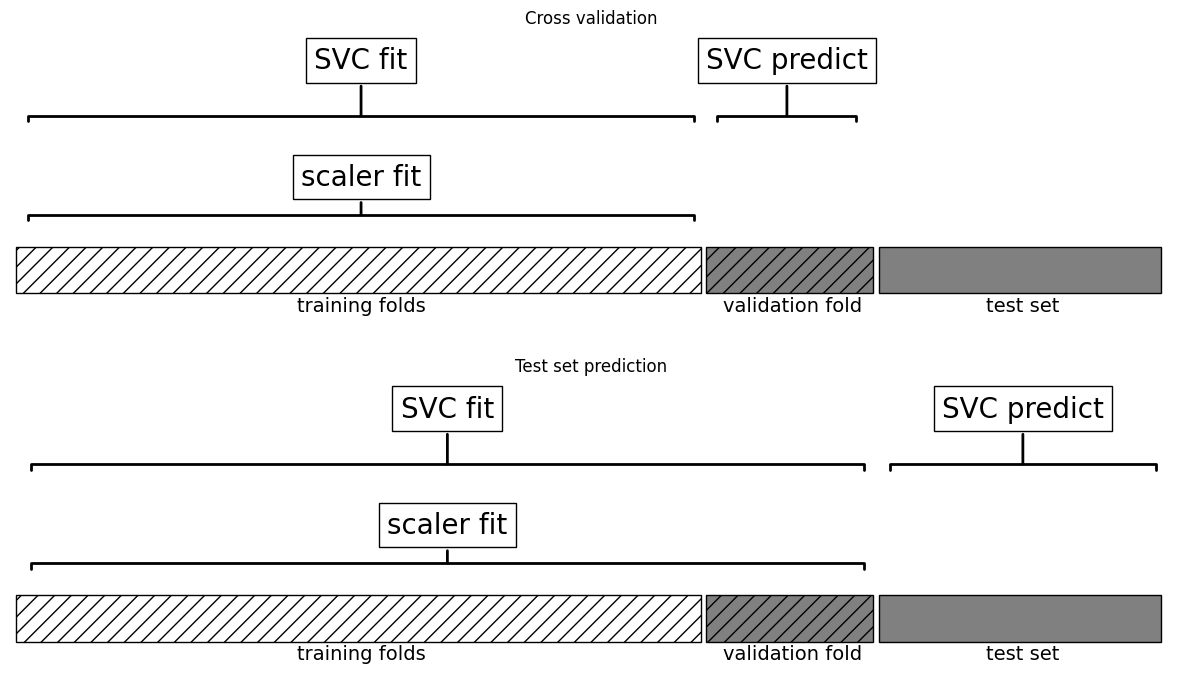
El impacto de la fuga de información en la validación cruzada depende del preprocesamiento. La estimación de escala en el pliegue de prueba tiene un impacto menor, pero usarlo en la extracción o selección de características puede distorsionar significativamente los resultados.
A diferencia de la búsqueda en red que hicimos antes, ahora para cada división en la validación cruzada, el
MinMaxScalerse reajusta solo con las divisiones de entrenamiento y no se filtra información de la división de prueba en la búsqueda de parámetros`
mglearn.plots.plot_proper_processing()
Veamos cómo podemos utilizar la clase
Pipelinepara expresar el flujo de trabajo para entrenar unaSVMdespués de escalar los datos conMinMaxScaler(por ahora sin grid-search). Primero, construimos un objeto pipeline proporcionándole una lista de pasos. Cada paso es una tupla que contiene un nombre (cualquier cadena de su elección) y una instancia de un estimadorEl uso de
PipelineenGridSearchCVes como con cualquier estimador: se define un diccionario de parámetros donde cada clave indica el paso y el parámetro usando doble guión bajo, por ejemplo,"svm__C"y"svm__gamma"si el modeloSVCse llama"svm"en elpipeline.
pipe = Pipeline([
("scaler", MinMaxScaler()),
("svm", SVC(probability=True))
])
param_grid_pipe = {
'svm__C': [0.01, 0.1, 1, 10, 100],
'svm__gamma': [0.01, 0.1, 1, 10, 100]
}
grid_pipe = GridSearchCV(pipe, param_grid=param_grid_pipe, cv=5, scoring="roc_auc")
grid_pipe.fit(X_train, y_train)
GridSearchCV(cv=5,
estimator=Pipeline(steps=[('scaler', MinMaxScaler()),
('svm', SVC(probability=True))]),
param_grid={'svm__C': [0.01, 0.1, 1, 10, 100],
'svm__gamma': [0.01, 0.1, 1, 10, 100]},
scoring='roc_auc')In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook. On GitHub, the HTML representation is unable to render, please try loading this page with nbviewer.org.
GridSearchCV(cv=5,
estimator=Pipeline(steps=[('scaler', MinMaxScaler()),
('svm', SVC(probability=True))]),
param_grid={'svm__C': [0.01, 0.1, 1, 10, 100],
'svm__gamma': [0.01, 0.1, 1, 10, 100]},
scoring='roc_auc')Pipeline(steps=[('scaler', MinMaxScaler()), ('svm', SVC(probability=True))])MinMaxScaler()
SVC(probability=True)
En el paso anterior, hemos creado dos pasos: el primero, llamado
"scaler", es una instancia de MinMaxScaler y el segundo, llamado"svm", es una instancia de SVC. Ahora, podemos ajustar nuestro pipeline, como cualquier otro estimador descikit-learn.grid_pipe.fitprimero llama afiten el primer paso (el escalador), luego transforma los datos de entrenamiento usando el escalador, y finalmente ajusta laSVMcon los datos escalados. Para evaluar en los datos de prueba, simplemente llamamos agrid_pipe.predict_proba
y_scores_pipe = grid_pipe.predict_proba(X_test)[:, 1]
auc_pipe = roc_auc_score(y_test, y_scores_pipe)
print("AUC sin data leakage (con pipeline): {:.3f}".format(auc_pipe))
print("Mejores parámetros (sin leakage):", grid_pipe.best_params_)
AUC sin data leakage (con pipeline): 0.500
Mejores parámetros (sin leakage): {'svm__C': 0.01, 'svm__gamma': 0.01}
El impacto de la fuga de información en la validación cruzada depende del preprocesamiento. La estimación de escala en el pliegue de prueba tiene un impacto menor, pero usarlo en la extracción o selección de características puede distorsionar significativamente los resultados.
10.3. Data Leakage#
La fuga de datos (data-leakage) ocurre cuando se utiliza información no disponible en el momento de la predicción, lo que produce estimaciones optimistas y un bajo rendimiento en producción. Suele originarse por una separación incorrecta entre entrenamiento y prueba, permitiendo que el modelo acceda, directa o indirectamente, a datos reservados.
Las transformaciones de preprocesamiento, como la normalización o la selección de características, deben ajustarse exclusivamente con los datos de entrenamiento; incluir los de prueba introduce sesgo. Esta situación se ilustrará mediante un problema de clasificación binaria con 10,000 características aleatorias.
import numpy as np
n_samples, n_features, n_classes = 200, 10000, 2
rng = np.random.RandomState(42)
X = rng.standard_normal((n_samples, n_features))
y = rng.choice(n_classes, n_samples)
Forma incorrecta
El uso de todos los datos para la selección de características genera una precisión artificialmente alta, incluso cuando
Xeyson independientes, lo que debería dar una precisión cercana a 0.5. Esto ocurre porque la selección de características “ve” los datos de prueba, otorgando una ventaja injusta.En el enfoque incorrecto, se seleccionan características antes de dividir los datos, lo que infla la precisión del modelo.
SelectKBestelige laskmejores características según una función de puntuación(X, y), reteniendo aquellas con mayor relación cony. Si se usachi2, se evalúa la dependencia entre cada característica ey: valores bajos indican independencia, valores altos sugieren relación no aleatoria. Por defecto,SelectKBestempleaf_regression F-valuepara medir la relevancia de cada característica.
from sklearn.model_selection import train_test_split
from sklearn.feature_selection import SelectKBest
from sklearn.ensemble import GradientBoostingClassifier
from sklearn.metrics import accuracy_score
Preprocesamiento incorrecto: se transforman todos los datos
X_selected = SelectKBest(k=25).fit_transform(X, y) #(Dependencia entre cada X[:,i] e y.) -> Reg: F de ANOVA o Class: Chi-cuadrado
X_train, X_test, y_train, y_test = train_test_split(X_selected, y, random_state=42)
gbc = GradientBoostingClassifier(random_state=1)
gbc.fit(X_train, y_train)
y_pred = gbc.predict(X_test)
accuracy_score(y_test, y_pred)
0.76
Forma correcta
Para evitar la fuga de datos, primero se deben dividir los datos en entrenamiento y prueba. La selección de características debe realizarse solo con el conjunto de entrenamiento. Al usar
fitofit_transform, se aplica exclusivamente al entrenamiento. Esto garantiza que la puntuación refleje el desempeño real del modelo.
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=42)
select = SelectKBest(k=25)
X_train_selected = select.fit_transform(X_train, y_train)
gbc = GradientBoostingClassifier(random_state=1)
gbc.fit(X_train_selected, y_train)
X_test_selected = select.transform(X_test)
y_pred = gbc.predict(X_test_selected)
accuracy_score(y_test, y_pred)
0.46
Se recomienda usar un pipeline para encadenar la selección de características y los estimadores, asegurando que solo los datos de entrenamiento se usen en el ajuste y los de prueba solo para evaluar la precisión.
from sklearn.pipeline import make_pipeline
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=42)
pipeline = make_pipeline(SelectKBest(k=25), GradientBoostingClassifier(random_state=1))
pipeline.fit(X_train, y_train)
y_pred = pipeline.predict(X_test)
accuracy_score(y_test, y_pred)
0.46
10.4. Ilustración de la fuga de datos#
Un ejemplo clásico de fuga de información en validación cruzada se encuentra en The Elements of Statistical Learning de Hastie, Tibshirani y Friedman. Reproducimos una versión adaptada: en una tarea de regresión sintética con 100 muestras y 1.000 características, todas se generan de una distribución gaussiana independiente, al igual que la variable respuesta.
import numpy as np
rnd = np.random.RandomState(seed=0)
X = rnd.normal(size=(100, 10000))
y = rnd.normal(size=(100,))
No hay relación entre los datos \(X\) y el objetivo \(y\) (son independientes), por lo que no debería ser posible aprender nada del conjunto. Se selecciona la característica más informativa con
SelectPercentile(elige variables en el percentil más alto de scores) y se evalúa un regresorRidgemediante validación cruzada.
from sklearn.feature_selection import SelectPercentile, f_regression
select = SelectPercentile(score_func=f_regression, percentile=5).fit(X, y)
X_selected = select.transform(X)
print("X_selected.shape: {}".format(X_selected.shape))
X_selected.shape: (100, 500)
from sklearn.model_selection import cross_val_score
from sklearn.linear_model import Ridge
cross_val_score(Ridge(), X_selected, y, cv=5)
array([0.84834054, 0.94084243, 0.88541709, 0.94012139, 0.91425508])
print("Cross-validation accuracy (cv only on ridge): {:.2f}".format(np.mean(cross_val_score(Ridge(), X_selected, y, cv=5))))
Cross-validation accuracy (cv only on ridge): 0.91
El score de validación cruzada es 0.91, lo que sugiere un modelo excelente, pero esto es erróneo, ya que los datos son aleatorios. La selección de características eligió, por azar, algunas altamente correlacionadas con el objetivo. Como la selección ocurrió fuera de la validación cruzada, filtró información de los pliegues de prueba, generando resultados poco realistas. Para evitar esto, es necesario aplicar una validación cruzada adecuada con una tubería.
pipe = Pipeline([("select", SelectPercentile(score_func=f_regression, percentile=5)),
("ridge", Ridge())])
cross_val_score(pipe, X, y, cv=5)
array([-0.97502994, -0.03166358, -0.03989415, 0.03018385, -0.2163673 ])
print("Cross-validation accuracy (pipeline): {:.2f}".format(np.mean(cross_val_score(pipe, X, y, cv=5))))
Cross-validation accuracy (pipeline): -0.25
La API de scoring siempre maximiza la puntuación. Por ello, las métricas a minimizar (como MSE) se devuelven en negativo, mientras que las que deben maximizarse se mantienen en positivo.
Pipeline garantiza que la selección de características ocurra dentro de la validación cruzada, evitando la fuga de datos. Esto impide que el modelo seleccione características basadas en información del conjunto de prueba, lo que podría generar resultados engañosos.
10.5. Reduccción de dimensionalidad en el Pipeline#
En tareas de aprendizaje supervisado con datos de alta dimensionalidad, la reducción de dimensionalidad puede ser fundamental para mejorar la eficiencia computacional y evitar el sobreajuste. Una técnica habitual es la aplicación de PCA (Análisis de Componentes Principales) como paso previo al modelado. Para evitar data leakage, estas transformaciones deben integrarse correctamente en un flujo de trabajo reproducible.
A continuación, se muestra un ejemplo con el conjunto de datos
digitsdonde se implementa unPipelineque incluye:Estandarización (
StandardScaler),Reducción de dimensionalidad (
PCA),Clasificación (
KNeighborsClassifier),
junto con una búsqueda de hiperparámetros usando
GridSearchCV.
from sklearn.decomposition import PCA
from sklearn.neighbors import KNeighborsClassifier
from sklearn.pipeline import Pipeline
from sklearn.model_selection import GridSearchCV
from sklearn.preprocessing import StandardScaler
from sklearn import datasets
import numpy as np
import matplotlib.pyplot as plt
# ==============================
# 1. Datos de ejemplo
# ==============================
X_digits, y_digits = datasets.load_digits(return_X_y=True)
# ==============================
# 2. Definimos el pipeline
# ==============================
pipe = Pipeline(steps=[
("scaler", StandardScaler()),
("pca", PCA()),
("knn", KNeighborsClassifier())
])
# ==============================
# 3. Parámetros para GridSearchCV
# ==============================
param_grid = {
"pca__n_components": [5, 15, 30, 45, 60],
"knn__n_neighbors": [3, 5, 7, 9],
"knn__weights": ["uniform", "distance"]
}
# ==============================
# 4. Búsqueda de hiperparámetros
# ==============================
search = GridSearchCV(pipe, param_grid, n_jobs=-1)
search.fit(X_digits, y_digits)
print(f"Mejor puntuación CV: {search.best_score_:.3f}")
print("Mejores parámetros encontrados:", search.best_params_)
# ==============================
# 5. Extraer PCA y modelo final
# ==============================
best_pca = search.best_estimator_.named_steps["pca"]
# Varianza explicada en proporción
varianza = best_pca.explained_variance_ratio_
# Mostrar las 10 componentes más importantes según PCA
top10 = np.argsort(varianza)[::-1][:10]
print("\nTop 10 componentes PCA por varianza explicada:")
for i in top10:
print(f"Componente {i+1}: {varianza[i]:.4f}")
# ==============================
# 6. Visualización del espectro PCA
# ==============================
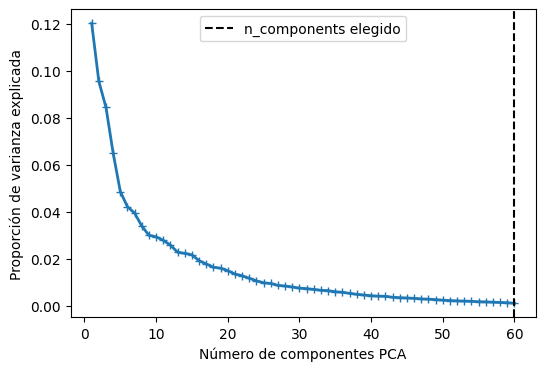
plt.figure(figsize=(6, 4))
plt.plot(np.arange(1, best_pca.n_components_ + 1),
varianza, marker="+", linewidth=2)
plt.axvline(search.best_estimator_.named_steps["pca"].n_components,
color='k', linestyle='--', label="n_components elegido")
plt.ylabel("Proporción de varianza explicada")
plt.xlabel("Número de componentes PCA")
plt.legend()
plt.show()
Mejor puntuación CV: 0.948
Mejores parámetros encontrados: {'knn__n_neighbors': 3, 'knn__weights': 'distance', 'pca__n_components': 60}
Top 10 componentes PCA por varianza explicada:
Componente 1: 0.1203
Componente 2: 0.0956
Componente 3: 0.0844
Componente 4: 0.0650
Componente 5: 0.0486
Componente 6: 0.0421
Componente 7: 0.0394
Componente 8: 0.0339
Componente 9: 0.0300
Componente 10: 0.0293
Observación
Cuando se entrena un pipeline \(\textsf{StandardScaler}() \Longrightarrow \textsf{PCA}() \Longrightarrow \textsf{KNeighborsClassifier}()\) de la siguiente forma:
pipeline.fit(X_train, y_train)
Ocurre lo siguiente en orden:
Paso PCA:
El PCA calculó las componentes principales usando
X_train.Se quedó con las primeras 60 (por ejemplo) y guardó la matriz de proyección.
Paso KNN:
Recibió las proyecciones reducidas de
X_train(60 columnas en vez de miles).Guardó la posición de cada punto de entrenamiento en ese nuevo espacio.
No aprendió “coeficientes”, solo almacenó los puntos (porque KNN es un algoritmo de memoria).
Cuando llega un nuevo dataset de test
y_pred = pipeline.predict(X_test)
El pipeline hace automáticamente las mismas transformaciones que usó en entrenamiento, pero sin volver a recalcular nada:
Paso PCA:
Aplica la misma proyección aprendida con
X_trainpara transformarX_test. Es decir, no vuelve a buscar nuevas componentes, usa las mismas 60 direcciones ya aprendidas.Resta la misma media que usó con
X_train. ProyectaX_testsobre las mismas 60 direcciones que aprendió antes.Esto asegura que
X_testquede en el mismo espacio de características queX_train.
Paso KNN:
Calcula la distancia entre cada punto transformado de
X_testy todos los puntos deX_trainen ese espacio reducido.Busca los k vecinos más cercanos y asigna la clase según
weights:"uniform"→ todos los vecinos pesan igual."distance"→ vecinos más cercanos pesan más en la votación.
10.6. La interfaz general del Pipeline#
La clase
Pipelineno se limita al preprocesamiento y la clasificación, sino que puede combinar múltiples estimadores, como extracción de características, selección, escalado y clasificación, en una secuencia de pasos. También puede incluir regresión o agrupación en lugar de clasificación. El único requisito es que todos los pasos, excepto el último, deben tener un métodotransformpara encadenar su salida con el siguiente paso.Durante
Pipeline.fit, cada paso aplicafitytransform, excepto el último, que solo usafit. Internamente, la tubería ejecuta los métodos en secuencia, conpipeline.steps[i][1]representando cada estimador en la lista de pasos.
def fit(self, X, y):
X_transformed = X
for name, estimator in self.steps[:-1]:
# Iterar sobre todo excepto el último paso
# Ajustar y transformar los datos
X_transformed = estimator.fit_transform(X_transformed, y)
# Ajuste en el último paso
self.steps[-1][1].fit(X_transformed, y)
return self
Cuando se predice utilizando
Pipeline, transformamos los datos de forma similar utilizando todos los pasos menos el último paso, y luego llamamos apredicten el último paso
def predict(self, X):
X_transformed = X
for step in self.steps[:-1]:
# Iterar sobre todo excepto el último paso
# Ajustar y transformar los datos
X_transformed = step[1].transform(X_transformed)
# Ajuste en el último paso
return self.steps[-1][1].predict(X_transformed)
El proceso se ilustra en la Fig. 10.1 para dos transformadores,
T1,T2, y unclassifier(llamadoClassifier)
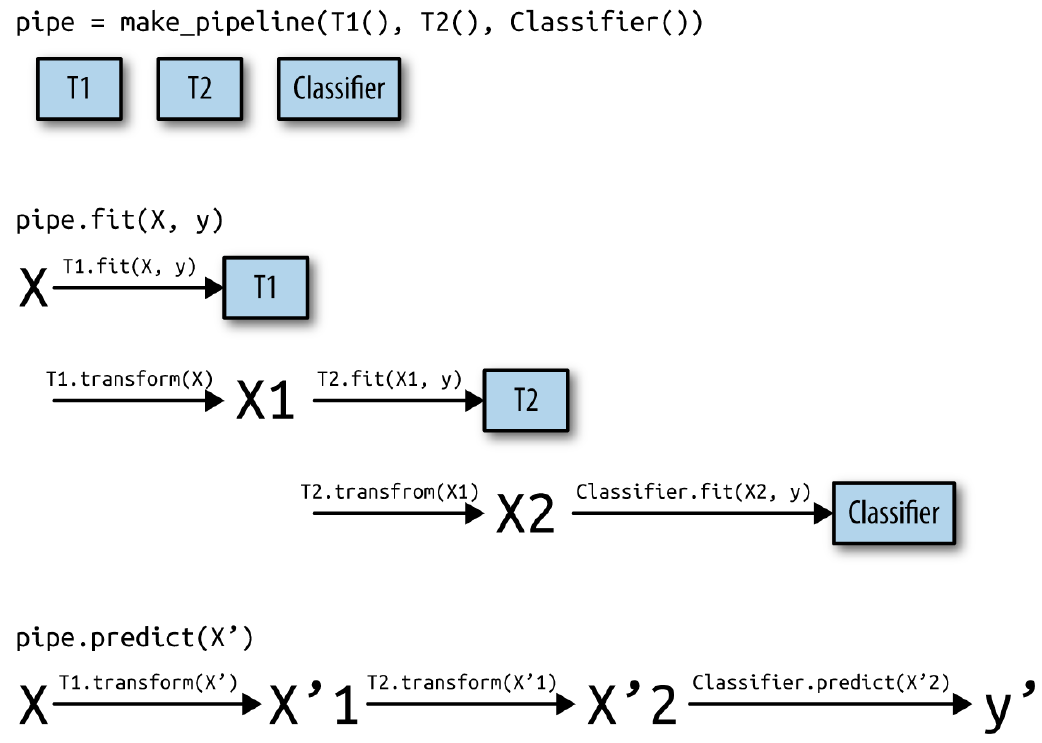
Fig. 10.1 Visión general del proceso de entrenamiento y predicción de la tubería.#
Un
Pipelinees más general de lo que parece: no es necesario que el último paso incluya un modelo con función de predicción. Puede componerse solo de transformaciones, como un escalador seguido de un PCA. En ese caso, si el último paso tiene un métodotransform, elPipelinetambién podrá aplicartransformtras procesar los datos en cada etapa previa. Lo único indispensable es que el paso final tenga un métodofit.
10.7. Creación cómoda de pipelines con make_pipeline#
La creación de un
Pipelineutilizando la sintaxis descrita anteriormente es a veces un poco engorrosa, y a menudo no necesitamos nombres especificados por el usuario para cada paso. Existe una función convenientemake_pipeline, que creará una tubería por nosotros y nombrará automáticamente cada paso basándose en su clase. La sintaxis demake_pipelinees la siguiente
from sklearn.pipeline import make_pipeline
pipe_long = Pipeline([("scaler", MinMaxScaler()), ("svm", SVC(C=100))])
pipe_short = make_pipeline(MinMaxScaler(), SVC(C=100))
Los objetos pipeline
pipe_longypipe_shorthacen exactamente lo mismo, peropipe_shorttiene pasos que fueron nombrados automáticamente. Podemos ver los nombres de los pasos mirando el atributosteps
print("Pipeline steps:\n{}".format(pipe_short.steps))
Pipeline steps:
[('minmaxscaler', MinMaxScaler()), ('svc', SVC(C=100))]
Los pasos se denominan
minmaxscalerysvc. En general, los nombres de los pasos son sólo versiones de los nombres de las clases. Si varios pasos tienen la misma clase, se añade un número
from sklearn.preprocessing import StandardScaler
from sklearn.decomposition import PCA
pipe = make_pipeline(StandardScaler(), PCA(n_components=2), StandardScaler())
print("Pipeline steps:\n{}".format(pipe.steps))
Pipeline steps:
[('standardscaler-1', StandardScaler()), ('pca', PCA(n_components=2)), ('standardscaler-2', StandardScaler())]
Como puede ver, el primer paso de
StandardScalerllamóstandardscaler-1y el segundostandardscaler-2. Sin embargo, en este tipo de configuraciones podría ser mejor utilizar la construcción de tuberías con nombres explícitos, para dar nombres más semánticos a cada paso
10.8. Acceso a los atributos de los pasos#
Para inspeccionar atributos de pasos en un
Pipeline, como coeficientes de un modelo o componentes dePCAse utiliza el atributonamed_steps, un diccionario que vincula nombres de pasos con sus estimadores. Tras ajustar elpipeline, se pueden extraer, por ejemplo, las dos primeras componentes principales del pasopcaaplicado al conjunto de datoscancer.
from sklearn.datasets import load_breast_cancer
cancer = load_breast_cancer()
X = cancer.data
y = cancer.target
pipe.fit(X, y)
components = pipe.named_steps["pca"].components_
print("components.shape: {}".format(components.shape))
components.shape: (2, 30)
10.9. Acceso a los atributos en un Pipeline con GridSearch#
Como hemos comentado anteriormente en este capítulo, una de las principales razones para utilizar
pipelines, es para realizar búsquedas en la red. Una tarea común es acceder a algunos de los pasos de una tubería dentro de dentro de una búsqueda en red.Busquemos en la red un clasificador
LogisticRegressionen el conjunto de datoscancer, utilizandoPipeline. UsamosStandardScaler, para escalar los datos antes de pasarlos al clasificadorLogisticRegresión. Primero creamos una instancia de nuestropipelineusando la funciónmake_pipeline.
from sklearn.linear_model import LogisticRegression
pipe = make_pipeline(StandardScaler(), LogisticRegression(max_iter=1000))
A continuación, creamos un grid de parámetros. Como se explica en la sección anterior, el parámetro de regularización para
LogisticRegressiones el parámetroC. Utilizamos una red logarítmica para este parámetro, buscando entre 0.01 y 100.Como utilizamos la función
make_pipeline, el nombre del pasoLogisticRegressionen elpipelinees el nombre de la clase en minúsculas,logisticregression. Para afinar el parámetroC, tenemos que especificar una red de parámetros paralogisticregression__C
param_grid = {'logisticregression__C': [0.01, 0.1, 1, 10, 100]}
Como es habitual, dividimos el conjunto de datos
canceren conjuntos de entrenamiento y de prueba, y ajustamos una búsqueda en red
X_train, X_test, y_train, y_test = train_test_split(cancer.data, cancer.target, random_state=4)
grid = GridSearchCV(pipe, param_grid, cv=5)
grid.fit(X_train, y_train)
GridSearchCV(cv=5,
estimator=Pipeline(steps=[('standardscaler', StandardScaler()),
('logisticregression',
LogisticRegression(max_iter=1000))]),
param_grid={'logisticregression__C': [0.01, 0.1, 1, 10, 100]})In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook. On GitHub, the HTML representation is unable to render, please try loading this page with nbviewer.org.
GridSearchCV(cv=5,
estimator=Pipeline(steps=[('standardscaler', StandardScaler()),
('logisticregression',
LogisticRegression(max_iter=1000))]),
param_grid={'logisticregression__C': [0.01, 0.1, 1, 10, 100]})Pipeline(steps=[('standardscaler', StandardScaler()),
('logisticregression', LogisticRegression(max_iter=1000))])StandardScaler()
LogisticRegression(max_iter=1000)
Entonces, ¿cómo accedemos a los coeficientes del mejor modelo
LogisticRegressionque fue encontrado porGridSearchCV? De los capítulos anteriores sabemos que el mejor modelo encontrado porGridSearchCV, entrenado con todos los datos de entrenamiento, se almacena engrid.best_estimator_
print("Best estimator:\n{}".format(grid.best_estimator_))
Best estimator:
Pipeline(steps=[('standardscaler', StandardScaler()),
('logisticregression', LogisticRegression(C=1, max_iter=1000))])
Este
best_estimator_en nuestro caso es unpipelinecon dos pasos,standardcalerylogisticregression. Para acceder al paso delogisticregression, podemos utilizar el atributonamed_stepsde la tubería, como se ha explicado anteriormente
print("Logistic regression step:\n{}".format(grid.best_estimator_.named_steps["logisticregression"]))
Logistic regression step:
LogisticRegression(C=1, max_iter=1000)
Ahora que tenemos la instancia de
LogisticRegressionentrenada, podemos acceder a los coeficientes (pesos) asociados a cada característica de entrada. Para mas información sobre los parámetros que podemos visualizar de este modelo clasificador (ver LogisticRegression). En este caso visualizaremos sus coeficientes usando.coef_. Puede que sea una expresión algo larga, pero a menudo resulta útil para entender los modelos.
print("Logistic regression coefficients:\n{}".format(grid.best_estimator_.named_steps["logisticregression"].coef_))
Logistic regression coefficients:
[[-0.43570655 -0.34266946 -0.40809443 -0.5344574 -0.14971847 0.61034122
-0.72634347 -0.78538827 0.03886087 0.27497198 -1.29780109 0.04926005
-0.67336941 -0.93447426 -0.13939555 0.45032641 -0.13009864 -0.10144273
0.43432027 0.71596578 -1.09068862 -1.09463976 -0.85183755 -1.06406198
-0.74316099 0.07252425 -0.82323903 -0.65321239 -0.64379499 -0.42026013]]
10.10. Pasos de preprocesamiento GridSearch y Parámetros del Modelo#
Usando
pipelines, podemos encapsular todos los pasos de procesamiento en nuestro flujo de trabajo de aprendizaje automático en un único estimador descikit-learn. Otro beneficio de hacer esto es que ahora podemosajustar los parámetros de preprocesamientousando el output de una tarea supervisada, como la regresión o la clasificación. En los capítulos anteriores, utilizamos las funciones polinómicas en el datasetbostonantes de aplicar elregresor ridge. Vamos a modelar esto usando un pipeline en su lugar. El proceso contiene tres pasos:escalar los datos, calcular las características polinómicas y la regresión ridge
import pandas as pd
data_url = "http://lib.stat.cmu.edu/datasets/boston"
raw_df = pd.read_csv(data_url, sep="\s+", skiprows=22, header=None)
data = np.hstack([raw_df.values[::2, :], raw_df.values[1::2, :2]])
target = raw_df.values[1::2, 2]
X_train, X_test, y_train, y_test = train_test_split(data, target, random_state=0)
from sklearn.preprocessing import PolynomialFeatures
pipe = make_pipeline(StandardScaler(), PolynomialFeatures(), Ridge())
PolynomialFeaturesgenera características polinómicas e interacciones hasta un grado especificado. Por ejemplo, para una entrada[a, b]y grado 2, se obtiene[1, a, b, a², ab, b²]. La elección del grado adecuado depende del desempeño del modelo. Se puede optimizar junto conalphadeRidgeusando unparam_griden un pipeline.
param_grid = {'polynomialfeatures__degree': [1, 2, 3],
'ridge__alpha': [0.001, 0.01, 0.1, 1, 10, 100]}
Ahora podemos volver a ejecutar nuestra búsqueda en la red utilizando nuestro
param_grid
grid = GridSearchCV(pipe, param_grid=param_grid, cv=5, n_jobs=-1)
grid.fit(X_train, y_train)
GridSearchCV(cv=5,
estimator=Pipeline(steps=[('standardscaler', StandardScaler()),
('polynomialfeatures',
PolynomialFeatures()),
('ridge', Ridge())]),
n_jobs=-1,
param_grid={'polynomialfeatures__degree': [1, 2, 3],
'ridge__alpha': [0.001, 0.01, 0.1, 1, 10, 100]})In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook. On GitHub, the HTML representation is unable to render, please try loading this page with nbviewer.org.
GridSearchCV(cv=5,
estimator=Pipeline(steps=[('standardscaler', StandardScaler()),
('polynomialfeatures',
PolynomialFeatures()),
('ridge', Ridge())]),
n_jobs=-1,
param_grid={'polynomialfeatures__degree': [1, 2, 3],
'ridge__alpha': [0.001, 0.01, 0.1, 1, 10, 100]})Pipeline(steps=[('standardscaler', StandardScaler()),
('polynomialfeatures', PolynomialFeatures()),
('ridge', Ridge())])StandardScaler()
PolynomialFeatures()
Ridge()
Podemos visualizar el resultado de la validación cruzada utilizando un mapa de calor (ver matshow), como ya se hizo en anteriores secciones
import matplotlib.pyplot as plt
plt.matshow(grid.cv_results_['mean_test_score'].reshape(3, -1), vmin=0, cmap="viridis")
plt.xlabel("ridge__alpha")
plt.ylabel("polynomialfeatures__degree")
plt.xticks(range(len(param_grid['ridge__alpha'])), param_grid['ridge__alpha'])
plt.yticks(range(len(param_grid['polynomialfeatures__degree'])),
param_grid['polynomialfeatures__degree'])
plt.colorbar();
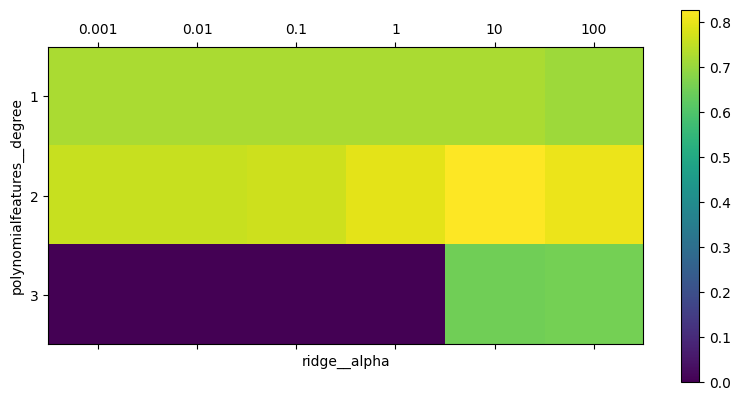
Observando los resultados producidos por la validación cruzada,
podemos ver que el uso de polinomios de grado dos ayuda, pero que los polinomios de grado tres son mucho peores que los de grado uno o dos. Esto se refleja en los mejores parámetros encontrados
print("Best parameters: {}".format(grid.best_params_))
Best parameters: {'polynomialfeatures__degree': 2, 'ridge__alpha': 10}
Lo que lleva al siguiente
score
print("Test-set score: {:.2f}".format(grid.score(X_test, y_test)))
Test-set score: 0.77
Hagamos una búsqueda en red sin características polinómicas para comparar
param_grid = {'ridge__alpha': [0.001, 0.01, 0.1, 1, 10, 100]}
pipe = make_pipeline(StandardScaler(), Ridge())
grid = GridSearchCV(pipe, param_grid, cv=5)
grid.fit(X_train, y_train)
print("Score without poly features: {:.2f}".format(grid.score(X_test, y_test)))
Score without poly features: 0.63
El no usar características polinómicas empeora los resultados. Buscar simultáneamente los parámetros del preprocesamiento y del modelo es una estrategia poderosa. Sin embargo,
GridSearchCVevalúa todas las combinaciones posibles, por lo que añadir más parámetros incrementa exponencialmente el número de modelos a construir.
10.11. Grid-Searching: ¿Qué modelo usar?#
GridSearchCVpuede combinarse conPipelinepara optimizar tanto los hiperparámetros como los pasos de preprocesamiento, como la elección entreStandardScaleroMinMaxScaler, lo cual amplía el espacio de búsqueda y debe aplicarse con cautela.Por ejemplo, al comparar
SVCyRandomForestClassifieren el conjuntoiris, se evalúa siSVCmejora con escalado, mientras queRandomForestno lo requiere. Para ello, se define unPipelinecon dos pasos: preprocesamiento y modelo, utilizandoSVCjunto conStandardScaler.
pipe = Pipeline([('preprocessing', StandardScaler()), ('classifier', SVC())])
Definimos
parameter_gridpara buscar entreRandomForestClassifierySVC, ajustando sus parámetros y preprocesamiento según el modelo. Usamos una lista de búsqueda para gestionar configuraciones distintas. Para asignar un estimador a un paso, usamos su nombre como parámetro. Si un modelo no requiere preprocesamiento (comoRandomForest), configuramos ese paso comoNone.
from sklearn.ensemble import RandomForestClassifier
param_grid = [{'classifier': [SVC()],
'preprocessing': [StandardScaler(), None],
'classifier__gamma': [0.001, 0.01, 0.1, 1, 10, 100],
'classifier__C': [0.001, 0.01, 0.1, 1, 10, 100]},
{'classifier': [RandomForestClassifier(n_estimators=100)],
'preprocessing': [None],
'classifier__max_features': [1, 2, 3]}]
Ahora podemos instanciar y ejecutar
grid searchcomo de costumbre, aquí en el conjunto de datoscancer. Nótes que solo es necesario inicializarpipecon cualquiera de los dos estimadores, luegoGridSearchCVse encargará de seleccionar el mejor modelo, basado en elparam_gridsuministrado.
X_train, X_test, y_train, y_test = train_test_split(cancer.data, cancer.target, random_state=0)
grid = GridSearchCV(pipe, param_grid, cv=5)
grid.fit(X_train, y_train)
print("Best params:\n{}\n".format(grid.best_params_))
print("Best cross-validation score: {:.2f}".format(grid.best_score_))
print("Test-set score: {:.2f}".format(grid.score(X_test, y_test)))
Best params:
{'classifier': SVC(C=10, gamma=0.01), 'classifier__C': 10, 'classifier__gamma': 0.01, 'preprocessing': StandardScaler()}
Best cross-validation score: 0.99
Test-set score: 0.98
El resultado de la búsqueda en la red es que el
SVCcon el preprocesamientoStandardScaler,C=10ygamma=0.01dio el mejor resultado de clasificación, en comparación del elRandomForestClassifier.
Resumen y conclusiones
La clase
Pipelinepermite encadenar múltiples pasos de procesamiento en un flujo de trabajo de aprendizaje automático, encapsulándolos en un solo objeto que sigue la interfaz descikit-learn(fit,predict,transform). Su uso es clave para una evaluación adecuada mediante validación cruzada y búsqueda en red.Además,
Pipelinesimplifica el código y minimiza errores, como olvidar aplicar transformaciones en el conjunto de prueba o desordenar los pasos. La elección de preprocesamiento y modelos requiere ensayo y error, peroPipelinefacilita la experimentación sin excesiva complejidad.
10.12. Proyecto Integrador de Aprendizaje Automático#
10.12.1. Contexto del Problema#
Las enfermedades cardiovasculares son la principal causa de muerte en todo el mundo, según la OMS. Detectarlas a tiempo es fundamental para evitar complicaciones graves. Con la digitalización de los historiales médicos y la disponibilidad de datos clínicos estructurados, ahora es posible utilizar técnicas de aprendizaje automático para predecir el riesgo de falla cardíaca.
El dataset “Heart Failure Prediction” contiene registros clínicos de pacientes, con variables como presión sanguínea, colesterol, glucosa, edad, entre otras. El objetivo es construir un modelo de clasificación binaria que prediga si un paciente está en riesgo de sufrir una falla cardíaca o no (HeartDisease = 1 o 0).
Este proyecto guía el desarrollo de un flujo completo de Machine Learning Operations (MLOps) de manera local, con herramientas modernas como FastAPI, Docker, Kubernetes, CI/CD con GitHub Actions y monitoreo con Evidently.
10.12.2. Instrucciones para el Desarrollo#
Descargar el dataset desde Kaggle: Heart Failure Prediction Dataset
Crear la estructura de carpetas propuesta en la Etapa 0.
Avanza por cada una de las etapas, desde el análisis inicial hasta el monitoreo del modelo en producción.
Puedes ejecutar todo localmente (sin necesidad de la nube), usando Docker y Minikube.
Cada etapa corresponde a un paso real en el ciclo de vida de un modelo de ML en producción.
10.12.3. Objetivo Final#
Desarrollar, evaluar y desplegar un modelo de predicción de falla cardíaca, con control de calidad y monitoreo integrado, aplicando prácticas de MLOps en tu entorno local.
10.12.4. Etapas del Proyecto#
Etapa |
Descripción |
|---|---|
0. Estructura |
Crear una estructura modular para alojar código, notebooks, APIs, despliegues y CI/CD. |
1. Análisis Exploratorio & Preprocesamiento |
Explorar el dataset, tratar valores nulos, evitar data leakage y preparar los datos. |
2. Entrenamiento Seguro |
Construir un pipeline con |
3. Despliegue Local (FastAPI + Docker) |
Servir el modelo mediante una API REST. Contenerizar la aplicación con Docker. |
4. Orquestación (Kubernetes) |
Crear manifiestos de Kubernetes ( |
5. Integración Continua (GitHub Actions) |
Agregar pruebas automáticas, revisión de estilo ( |
6. Monitoreo (Evidently) |
Generar reportes de deriva de datos (data drift) entre entrenamiento y predicción. |
10.12.5. ETAPA 0: Estructura de carpetas#
heart-disease-mlops/
├── app/
│ └── api.py
├── docker/
│ ├── Dockerfile
│ └── requirements.txt
├── k8s/
│ ├── deployment.yaml
│ └── service.yaml
├── notebooks/
│ ├── 1_model_leakage_demo.ipynb
│ └── 2_model_pipeline_cv.ipynb
├── .github/
│ └── workflows/
│ └── ci.yml
├── drift_report.html
├── model.joblib
└── README.md
10.12.6. ETAPA 1: Preprocesamiento y detección de Data Leakage#
Archivo: notebooks/1_model_leakage_demo.ipynb
Se utiliza el dataset de predicción de enfermedad cardíaca. Puede descargarse desde Kaggle o UCI Machine Learning Repository.
Ejemplo de flujo con y sin data leakage:
import pandas as pd
import numpy as np
from sklearn.model_selection import train_test_split, GridSearchCV
from sklearn.svm import SVC
from sklearn.preprocessing import MinMaxScaler
from sklearn.pipeline import Pipeline
from sklearn.metrics import roc_auc_score
# Cargar datos
df = pd.read_csv("heart.csv")
X = df.drop("target", axis=1)
y = df["target"]
# Variable artificial que introduce fuga
np.random.seed(0)
X["leaky_feature"] = y + np.random.normal(0, 0.01, size=len(y))
# Ejemplo con fuga de datos (data leakage)
scaler = MinMaxScaler()
X_scaled = scaler.fit_transform(X)
X_train_l, X_test_l, y_train_l, y_test_l = train_test_split(X_scaled, y, test_size=0.2, random_state=42)
grid_l = GridSearchCV(SVC(probability=True), param_grid={"C": [0.1, 1, 10], "gamma": [0.01, 0.1]}, cv=5, scoring="roc_auc")
grid_l.fit(X_train_l, y_train_l)
auc_l = roc_auc_score(y_test_l, grid_l.predict_proba(X_test_l)[:, 1])
# Flujo correcto (sin fuga)
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42)
pipe = Pipeline([("scaler", MinMaxScaler()), ("svc", SVC(probability=True))])
param_grid = {"svc__C": [0.1, 1, 10], "svc__gamma": [0.01, 0.1]}
grid = GridSearchCV(pipe, param_grid, cv=5, scoring="roc_auc")
grid.fit(X_train, y_train)
auc = roc_auc_score(y_test, grid.predict_proba(X_test)[:, 1])
print(f"AUC con fuga: {auc_l:.3f}")
print(f"AUC sin fuga: {auc:.3f}")
Implementar todos los modelos vistos hasta esta sección. Sugerencia: Agrega al menos tres clasificadores adicionales al flujo sin fuga (
RandomForest,KNN,LogisticRegression, etc.) utilizandoPipelineyGridSearchCV, y compara sus resultados (AUC) con los obtenidos por SVC.
Modelos recomendados:
LogisticRegressionRandomForestClassifierKNeighborsClassifierGradientBoostingClassifier
Requisitos:
Implementar cada modelo dentro de un
PipelineUtilizar
GridSearchCVpara optimizar hiperparámetrosComparar AUC y Accuracy
Presentar un ranking comparativo
Separar código en funciones reutilizables. Sugerencia: Encapsula la lógica de entrenamiento y evaluación en funciones claras y reutilizables.
Ejemplo:
def train_pipeline(X_train, y_train, model, param_grid):
pipe = Pipeline([("scaler", MinMaxScaler()), ("clf", model)])
grid = GridSearchCV(pipe, param_grid, cv=5, scoring="roc_auc")
grid.fit(X_train, y_train)
return grid
10.12.7. ETAPA 2: Modelado con validación segura#
Archivo: notebooks/2_model_pipeline_cv.ipynb
Este cuaderno debe contener:
División de los datos antes del escalado
Implementación de
PipelineconGridSearchCVEvaluación del modelo con matriz de confusión, curva ROC y AUC
10.12.8. ETAPA 3: Despliegue con FastAPI y Docker#
Archivo: app/api.py
API para predicciones en tiempo real:
from fastapi import FastAPI
from pydantic import BaseModel
import joblib
import numpy as np
model = joblib.load("app/model.joblib")
app = FastAPI()
class Input(BaseModel):
features: list
@app.post("/predict")
def predict(data: Input):
X = np.array(data.features).reshape(1, -1)
proba = model.predict_proba(X)[0][1]
return {"heart_disease_probability": proba, "prediction": int(proba > 0.5)}
Exportación del modelo:
import joblib
joblib.dump(grid.best_estimator_, "app/model.joblib")
Archivo: docker/Dockerfile
FROM python:3.10-slim
WORKDIR /app
COPY docker/requirements.txt .
RUN pip install --no-cache-dir -r requirements.txt
COPY app/ ./app/
CMD ["uvicorn", "app.api:app", "--host", "0.0.0.0", "--port", "8000"]
Archivo: docker/requirements.txt
fastapi
uvicorn
scikit-learn
joblib
pydantic
Construcción y ejecución local:
docker build -t heart-api -f docker/Dockerfile .
docker run -p 8000:8000 heart-api
10.12.9. ETAPA 4: Despliegue en Kubernetes local#
Archivo: k8s/deployment.yaml
apiVersion: apps/v1
kind: Deployment
metadata:
name: heart-model
spec:
replicas: 1
selector:
matchLabels:
app: heart-model
template:
metadata:
labels:
app: heart-model
spec:
containers:
- name: model
image: <TU_USUARIO_DOCKER>/heart-api
ports:
- containerPort: 8000
Archivo: k8s/service.yaml
apiVersion: v1
kind: Service
metadata:
name: heart-service
spec:
selector:
app: heart-model
ports:
- protocol: TCP
port: 80
targetPort: 8000
type: LoadBalancer
Comandos para desplegar:
kubectl apply -f k8s/deployment.yaml
kubectl apply -f k8s/service.yaml
kubectl get svc
10.12.10. ETAPA 5: Integración continua con GitHub Actions#
Archivo: .github/workflows/ci.yml
name: CI
on: [push]
jobs:
build:
runs-on: ubuntu-latest
steps:
- uses: actions/checkout@v3
- name: Set up Python
uses: actions/setup-python@v4
with:
python-version: '3.10'
- name: Install dependencies
run: |
pip install -r docker/requirements.txt
pip install flake8 pytest
- name: Lint
run: flake8 app/
- name: Tests
run: pytest tests/
10.12.11. ETAPA 6: Monitoreo de deriva de datos#
Uso de Evidently para monitoreo:
from evidently.report import Report
from evidently.metric_preset import DataDriftPreset
report = Report(metrics=[DataDriftPreset()])
report.run(reference_data=X_train, current_data=X_test)
report.save_html("drift_report.html")
10.12.12. README.md#
# Data Lake + Forecasting + Dashboard (MLOps local con Docker)
Este proyecto es una arquitectura local basada en herramientas open source y Docker para construir un **Data Lake local**, realizar **modelos de forecasting** y desplegar un **dashboard interactivo**. Está orientado a escenarios comerciales como análisis SELL IN / SELL OUT y toma de decisiones predictivas.
## Arquitectura y tecnologías utilizadas
- **MinIO**: almacenamiento de datos tipo S3 (Data Lake local)
- **Apache Spark**: procesamiento de datos y modelos de ML
- **Prophet (Facebook)**: modelos de forecasting de series temporales
- **Streamlit**: visualización interactiva
- **Airflow**: orquestación de pipelines
- **Docker**: contenedores para todas las herramientas
- **Jupyter Notebooks**: desarrollo y pruebas de los pasos
